Как расшифровывается ISO?
© 2015 Vasili-photo.com
Не секрет, что чувствительность фотоматериала измеряется в условных единицах ISO (см. «Экспозиция»). Это известно всем. Но что означает само слово «ISO»? Спросите любого фотолюбителя, и он с высокой вероятностью ответит вам, что ISO – это, мол, аббревиатура от International Standards Organization. Звучит правдоподобно, но это не совсем так. Точнее, это совсем не так.
Не существует такого учреждения, как International Standards Organization. Его попросту нет. Международная организация, занимающаяся разработкой стандартов в различных областях, называется International Organization for Standardization или, по-русски, Международная организация по стандартизации. Очевидно, что аббревиатурой для английского названия было бы IOS, а для русского и вовсе – МОС. Меж тем в технической литературе вовсю используются термины ISO или ИСО. Да и сами стандарты носят названия вроде ISO 12232:2006.
Так что же такое ISO? ISO или ИСО – это просто краткое название Международной организации по стандартизации. Не сокращённое, а именно краткое. Название ISO происходит от греческого слова ισος (isos), что значит равный. Такое название было выбрано для того, чтобы на любом языке мира оно звучало по возможности одинаково, в то время как аббревиатуры во всех языках получились бы разные. В этом можно убедиться на собственном сайте организации:
«Because ‘International Organization for Standardization’ would have different acronyms in different languages (IOS in English, OIN in French for Organisation internationale de normalisation), our founders decided to give it the short form ISO. ISO is derived from the Greek isos, meanin g equal. Whatever the country, whatever the language, we are always ISO».
К этому объяснению сложно что-нибудь добавить.
Таким образом, ISO – это не аббревиатура, как, например, AMD или IKEA, а просто название, как, например Fujifilm или Android. Вероятно, путаница возникает из-за того, что все буквы в слове ISO заглавные, вследствие чего, оно и вправду смахивает на аббревиатуру.
Вероятно, путаница возникает из-за того, что все буквы в слове ISO заглавные, вследствие чего, оно и вправду смахивает на аббревиатуру.
Кстати, слово ASA, используемое в старой (примерно до 1987 г.) литературе вместо ISO и означающее всё ту же чувствительность фотоплёнки, таки является настоящей аббревиатурой и расшифровывается как American Standards Association, т.е. Американский институт стандартов.
Спасибо за внимание!
Василий А.
Post scriptum
Если статья оказалась для вас полезной и познавательной, вы можете любезно поддержать проект, внеся вклад в его развитие. Если же статья вам не понравилась, но у вас есть мысли о том, как сделать её лучше, ваша критика будет принята с не меньшей благодарностью.
Не забывайте о том, что данная статья является объектом авторского права. Перепечатка и цитирование допустимы при наличии действующей ссылки на первоисточник, причём используемый текст не должен ни коим образом искажаться или модифицироваться.
Желаю удачи!
Дата публикации: 09.12.2013 |
Вернуться к разделу «Матчасть»
Перейти к полному списку статей
Что такое ISO?
ISO – это международная организация по стандартизации. Именно так в переводе на русский язык звучит ее полное название, по-английски: International Organization for Standardization. В русскоязычных источниках можно встретить как английскую аббревиатуру ISO, так и написание ИСО – это не ошибка, хотя на русский название организации не переводится с соблюдением тех же первых букв, что в английском варианте. Дело в том, что еще при создании организации стороны договорились использовать слово ИСО во всех языках без перевода, а лишь записывая его на местном алфавите. Русский язык, кстати сказать, наряду с английским и французским является одним из рабочих языков международной организации.
Основная функция ISO – организация работы смешанных экспертных групп, готовящих стандарты и руководства к ним в самых разных областях деятельности, от конкретных деталей для железнодорожного транспорта до таких сложных и многоаспектных проблем как экологическая безопасность на производстве, социальная ответственность бизнеса. Членами ISO являются не отдельные люди, а национальные организации по стандартизации. По правилам международной структуры, от каждой из стран может принимать участие в работе лишь один орган по стандартизации. Поэтому, фактически, они представляют интересы государств, хотя на бумаге или в уставных документах ISO это не закреплено. Впрочем, из правила есть исключения. Например, Гонконг, который имеет особый статус в составе Китая, представляет одна организация, а сам Китай – другая. Интересы России в ISO защищает Росстандарт (Федеральное агентство по техническому регулированию и метрологии, — ред.), но участники работы Международной организации по стандартизации – не всегда госорганы. В ряде других стран частные ассоциации и деловые объединения обладают таким статусом де факто, не имея никакого отношения к чиновничьему аппарату. Они заняли эту нишу в ходе естественного рыночного развития. С согласия правительств своих стран, они также могут выступать представителями в ISO.
В ряде других стран частные ассоциации и деловые объединения обладают таким статусом де факто, не имея никакого отношения к чиновничьему аппарату. Они заняли эту нишу в ходе естественного рыночного развития. С согласия правительств своих стран, они также могут выступать представителями в ISO.
Штаб-квартира ISO расположена в Женеве. Решения ISO принимаются консенсусом. В основном, они касаются выработки стандартов и других нормативных документов. Бизнес-сообщество и другие заинтересованные лица в каком-либо государстве сигнализируют национальному органу по стандартизации, что возникла настоятельная необходимость в создании нового нормативного документа. Тот, в свою очередь, имеет право поднять вопрос о создании стандарта на уровне ISO. Если другие участники работы структуры не возражают против этого, один из технических комитетов, то есть, экспертных групп, приступает к выработке необходимого документа. Значение ISO в российской деловой практике и в мировой, и отечественной стандартизации определяется тем, что это самая крупная, наиболее представительная и развитая международная организация в мире. Есть множество других объединений такого типа, но они далеко уступают ISO по своему развитию и преследуют цель создания востребованных нормативов в тех или иных отраслях, а не решение проблемы мировой гармонизации стандартов в принципе. Сегодня лишь ISO претендует на это. Интернациональные объединения государств, такие как ВТО или ЕС поощряют международную стандартизацию, так как она снимает торговые барьеры в многосторонних экономических отношениях.
Есть множество других объединений такого типа, но они далеко уступают ISO по своему развитию и преследуют цель создания востребованных нормативов в тех или иных отраслях, а не решение проблемы мировой гармонизации стандартов в принципе. Сегодня лишь ISO претендует на это. Интернациональные объединения государств, такие как ВТО или ЕС поощряют международную стандартизацию, так как она снимает торговые барьеры в многосторонних экономических отношениях.
На сегодняшний день, в ISO над новыми стандартами работают более 250 технических комитетов, уже разработано и опубликовано свыше 19 000 нормативных документов и других типов требований. Многие страны, в том числе и Россия, принимают переводы стандартов ISO на свой язык в качестве национальных стандартов. В общей сложности, это практикуют в 90 странах из 163 государств, которые имеют представительство в Международной организации по стандартизации. Некоторые стандарты Международной организации бесплатны, большая часть – находится в платном доступе.
Если вы нашли ошибку, пожалуйста, выделите фрагмент текста и нажмите Ctrl+Enter.
ИСО — это… Что такое ИСО?
ИСО
инженерно-строительный отдел
ИСО
Международная организация по стандартизации
англ.: ISO, International Organization for Standardization
англ., организация
Словарь: С. Фадеев. Словарь сокращений современного русского языка. — С.-Пб.: Политехника, 1997. — 527 с.
ИСО
инерциальная система отсчёта
ИСО
известково-серный отвар
фунгицид
Словарь: С. Фадеев. Словарь сокращений современного русского языка. — С.-Пб.: Политехника, 1997. — 527 с.
Фадеев. Словарь сокращений современного русского языка. — С.-Пб.: Политехника, 1997. — 527 с.
ИСО
информационно-справочный отдел
юр.
- ИО
- ИСО
исследование операций
- ИО
Источник: http://www.islu.ru/k_inform/glossary.html
ИСО
ипотечная специализированная организация
организация
Источник: http://ipocredit.ru/news/392356/
Пример использования
ОАО «ИСО ГПБ-Ипотека»
ИСО
инженерно-саперное отделение
воен.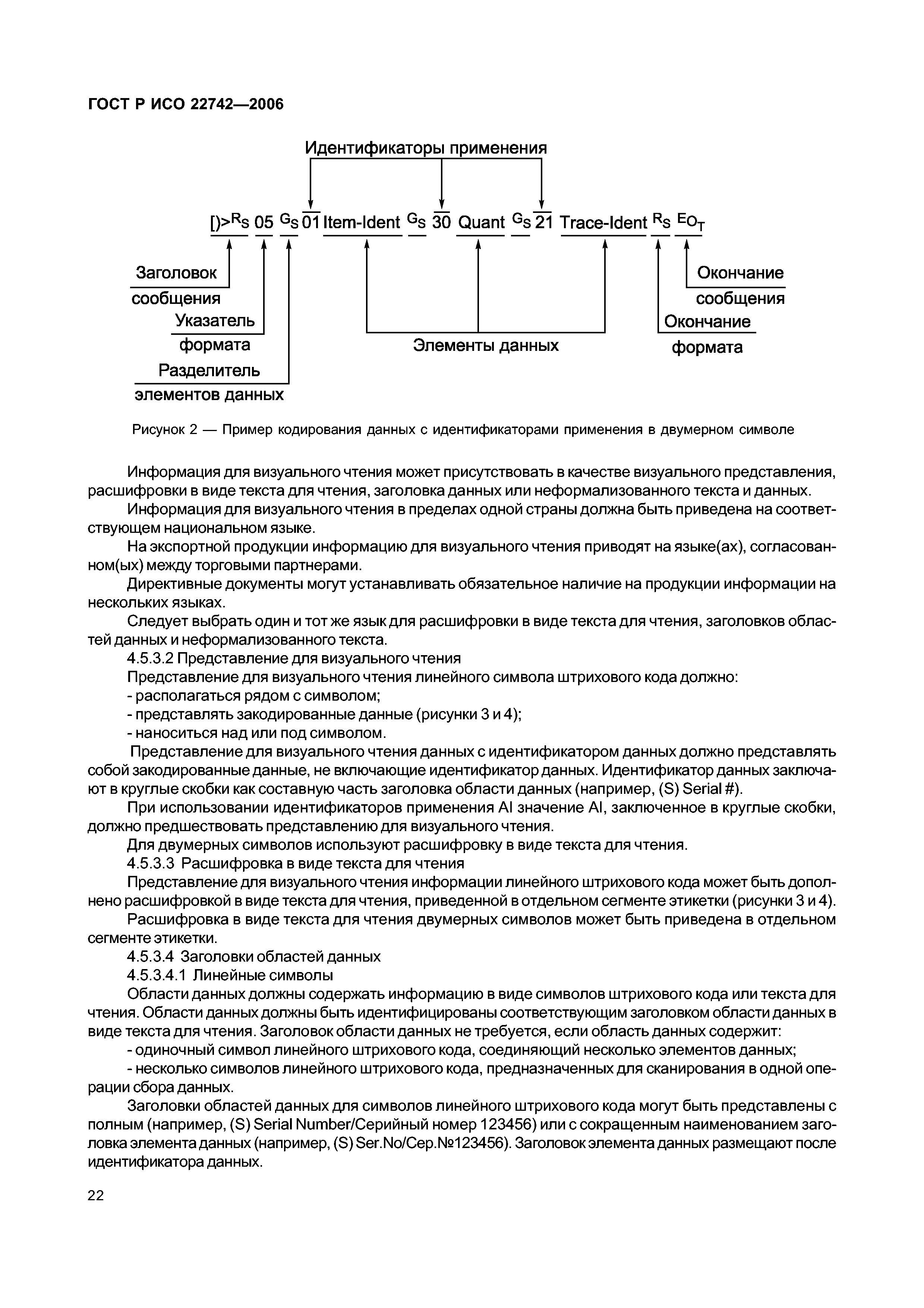
ИСО
интегрированная система охраны
ИСО
индикатор секторного обзора
Источник: Новый русско-английский политехнический словарь, ISBN 5-849347-01-6, 1998
ИСО
индивидуальный способ обучения
Источник: http://www.ug.ru/03.04/t5.htm
ИСО
Институт специального образования
УрГПУ
образование и наука
Источник: http://www.eduekb.ru/infm41. htm
htm
ИСО
«Информатизация системы образования»
программа Минобразования РФ
гос., образование и наука, РФ
Источник: http://e-rus.ru/news/2004/12/231537_16501.shtml
Словарь сокращений и аббревиатур.
Академик.
2015.
Стандарты ИСО | Журнал «Management» всё об ISO 9001
В условиях глобализации бизнеса и торговли, когда даже понятие «кризис» приобретает мировой характер, стремление к постоянному росту и развитию становится наиболее важным и первоочередным. Наиболее перспективные и быстро адаптирующиеся компании, способные проанализировать ситуацию и использовать ее в своих целях, смогут пережить смутные времена и реализовать свои амбиции.
В связи с этим в последнее время возникают вопросы о более эффективном, рациональном управлении, сертификации, о системах управления качеством, о стандарте ISO 9001. Попробуем разобраться в данных вопросах.
ЧТО ТАКОЕ ISO?
Аббревиатура ISO расшифровывается как International Organization for Standardization (Международная Организация по Стандартизации). Это организация, которая является всемирной федерацией национальных организаций по стандартизации.
В переводе с греческого слово «ΙΣΟΣ» (ИСОС) — «равный», вот почему на всех языках мира Международная Организация по Стандартизации имеет краткое название ISO (ИСО).
Участниками ISO сегодня являются более 150 стран мира, каждая из которых представлена национальными организациями по стандартизации. За период существования организации было опубликовано более 17 тысяч международных стандартов в различных областях деятельности. Начало было положено в период бурного подъема производства и, как следствие, мощного развития товарообмена между крупными индустриальными странами; внедрение стандартов приводило к удешевлению продукции, снижению затрат на производство, уменьшению отходов, повышению уровня совместимости продукции, произведенной в разных странах, упрощению ее использования и обслуживания.
ИСТОРИЯ ISO
Первые попытки международной стандартизации были предприняты в 1906г., когда была создана Международная электротехническая комиссия (МЭК) для унификации качества производимой продукции, которая была бы адаптирована к эксплуатации и признана потребителями различных странах мира. Данная международная стандартизация продукции явилась причиной создания в 1946 году международной федерации национальных ассоциаций по стандартизации, названной Международной Организацией по Стандартизации (ISO).
ISO была учреждена в Лондоне, где делегаты 25 стран приняли решение о возобновлении деятельности и создании организации, целью которой является обеспечение международной координации и унификации производственных стандартов.
В настоящее время Центральный секретариат ISO располагается в Женеве (Швейцария).
В 1987 году Международная Организация по Стандартизации выпустила серию норм по качеству, известных как серия ISO 9000, которые были разработаны для того, чтобы помочь организациям всех видов и размеров внедрить и обеспечить функционирование эффективных систем менеджмента, способствующих формированию на предприятиях культуры высокого качества.
КАКИЕ ПРЕИМУЩЕСТВА У МЕЖДУНАРОДНЫХ СТАНДАРТОВ ИСО?
Международные стандарты приносят технологические, экономические и социальные преимущества. Они помогают гармонизировать технические характеристики товаров и услуг, делают отрасль более эффективной и способствуют устранению барьеров в международной торговле. Соответствие международным стандартам помогает убедить потребителей, что продукты являются надежными, эффективными и безопасными для окружающей среды.
ДЛЯ БИЗНЕСА
Международные стандарты — это стратегические инструменты, руководства помогающие компаниям решить некоторые из самых насущных проблем современного бизнеса. Они обеспечивают как можно более высокую эффективность бизнес — операций, повышают производительность и помогают компаниям получить доступ на новые рынки.
Преимущества включают:
- Экономия расходов — международные стандарты позволяют оптимизировать операции и, тем самым, улучшают конечный результат
- Расширение потребительской удовлетворенности — стандарты помогают улучшить качество, повысить удовлетворенность потребителей и увеличить продажи
- Доступ к новым рынкам — стандарты помогают преодолеть торговые барьеры и открывают доступ на мировые рынки
- Увеличение доли рынка — стандарты помогают увеличить производительность и конкурентоспособность
- Экологические преимущества — стандарты помогают снизить негативное воздействие на окружающую среду
Преимущества в цифрах
- 2,5 млрд.
 фунтов стерлингов — годовой вклад стандартов в экономику Великобритании
фунтов стерлингов — годовой вклад стандартов в экономику Великобритании - 80% — доля влияния международных стандартов в мировой торговле
- 100 миллион. австр. долл. — выгода для экономики Австралии от выборочных стандартов в горнодобывающей промышленности
- 84% снижения сроков транспортировки благодаря контейнерным перевозкам
Стандарт iso 9000:2005 «системы менеджмента качества. Основные положения и словарь»
Стандарт iso 9001:2008 «системы менеджмента качества. Требования»
Стандарт iso 9004:2000 «системы менеджмента качества. Рекомендации по улучшению деятельности»
Стандарт iso 14001:2004 «системы экологического менеджмента. Требования и руководство по их применению»
Стандарт ohsas 18001:2007 «системы менеджмента охраны здоровья и обеспечения безопасности труда. Требования»
Требования»
Стандарт iso 19011:2002 «руководящие указания по проведению аудитов систем менеджмента качества и/или систем менеджмента окружающей среды»
Стандарт iso 22000:2005 «системы менеджмента безопасности пищевых продуктов. Требования ко всем организациям в цепи производства и потребления пищевых продуктов»
Техническая спецификация iso/ts 16949:2002 «системы менеджмента качества. Особые требования по применению исо 9001:2000 для организаций-производителей серийных и запасных частей для автомобильной промышленности»
Стандарт iso 17025:2005 «общие требования к компетентности испытательных и калибровочных лабораторий»
Стандарт iso 27001:2005 «системы управления информационной безопасностью. Требования»
Техническая спецификация iso/ts 29001:2003 «нефтяная, нефтехимическая и газовая отрасли промышленности. Отраслевые системы менеджмента качества. Требования к организациям, поставляющим продукцию и услуги»
Отраслевые системы менеджмента качества. Требования к организациям, поставляющим продукцию и услуги»
Стандарт iso/ tr 10013: 2001 «рекомендации по документированию систем менеджмента качества»
Стандарт iso/tr 10017:2003 «руководство по статистическим методам применительно к iso 9001:2000»
Стандарт iso 10019:2005 «руководящие указания по выбору консультантов системы менеджмента качества и использованию их услуг»
Что такое сертификат ИСО | ИСО: расшифровка и для чего нужно
Использование стандартов ISO пользуется все большей популярностью в мире. Так что такое ИСО, стандарт ISO или сертификат ИСО? ИСО – это аббревиатура, значение которой рассматривается в предлагаемом материале.
Что такое сертификат ISO
Под термином ISO понимают латинскую расшифровку (International Organization for Standardization) наименования организации, устанавливающей международные стандарты. Часто также можно встретить словосочетание «международный стандарт ISO». Общее количество этих стандартов превышает 23 тысячи, и каждый из них обозначается числовым кодом, который предваряет эта аббревиатура.
Часто также можно встретить словосочетание «международный стандарт ISO». Общее количество этих стандартов превышает 23 тысячи, и каждый из них обозначается числовым кодом, который предваряет эта аббревиатура.
Сертифицированные компании могут использовать знак ISO, который указывает, что они соответствуют требованиям стандарту.
Виды сертификатов ISO
Различают следующие виды сертификатов, с соответствующими числовыми обозначениями после стандартной аббревиатуры ISO:
- 9000 – устанавливает требования к качеству продукции;
- 14000 – экологическая безопасность;
- 22000 – безопасность продукции пищевого назначения;
- IEC 27000 – относительно информационной защиты;
- 10303 и 13584 – по управлению продукцией;
- 13485 – требования качества к изделиям медицинской сферы.
Сертификаты присваиваются претендентам по результатам анализа их соответствия предъявляемым требованиям.
Документы ИСО
Что касается документов, то ISO разрабатывает следующие типы документов: Стандарты ИСО (ISO Standards), Общедоступные технические требования (ISO/PAS), Технические требования (ISO/TS), Технические отчеты (ISO/TR), Соглашения международного семинара (ISO/IWA) и Руководства ИСО (ISO Guides).
Как понять, нужен ли вашей компании сертификат ИСО
Если компания стремится к расширению сферы деятельности, с привлечением международных партнеров, наличие сертификата, подтверждающего соответствие установленным стандартам, входит в число обязательных приоритетов.
В большинстве отраслей прохождение этой процедуры является добровольным. Но для предприятий, работающих в области пищевой промышленности, военного производства, авиастроении, наличие такого подтверждения качества продукции входит в число обязательных условий.
Преимущества при получении ISO 9001
Сертификат ISO 9001 обеспечивает достижение следующих конкурентных преимуществ:
- улучшения качества продукции;
- оптимизации и систематизации управленческого аппарата;
- повышения привлекательности имиджа;
- превосходства над конкурентами;
- облегчения процедуры лицензирования;
- возможности привлечения серьезных компаньонов;
- развития отношений с международными партнерами.

Успешное прохождение сертификации – первый и обязательный шаг на пути к международной интеграции компании. Организации, соответствующей этим требованиям, всегда отдается предпочтение при проведении тендерных торгов. Сертифицирующая компания определяет последующие пути развития бизнеса и указывает на слабые места, требующие улучшения.
По результатам изучения статистических исследований, проведенных иностранными экономистами, организации, успешно прошедшие сертификацию, улучшили эффективность работы управленческого аппарата, организацию персонала и степень вовлеченности работников в производственный процесс.
Перечисленные достоинства получения этих подтверждений соответствия международным требованиям обеспечивают увеличение общей прибыли компании до 10 процентов.
Проверка не ограничивается выдачей разрешительного документа. После сертификации, претендент проходит процедуру периодических аудитов, подтверждающих поддержание достигнутого уровня на необходимой отметке. Этот аудит выполняет тот же исполнитель, который сертифицировал заказчика.
Этот аудит выполняет тот же исполнитель, который сертифицировал заказчика.
Кто выдает сертификат ISO
Сертификат ИСО вправе выдать организация, прошедшая соответствующую аккредитацию в комитете, учредившем эти стандарты и устанавливающего образцы для следования предъявляемым требованиям качества.
Процедура сертификации предполагает последовательные этапы, предполагающие пересмотр и изменение следующей документации:
- основной, используемой внутри компании – включает распоряжения и приказы по предприятию, внутренний регламент качества продукции;
- вспомогательной для внутреннего применения – касается пересмотра нормативных, технологических и рабочих инструкций;
- внешней – об изменении отраслевых, государственных и международных стандартов, технологических и нормативных регламентов.
Выбор направления сертификации поможет сделать сертификационная организация, после изучения представленной заказчиком анкеты.
Не следует недооценивать важности получения сертификационного документа ИСО. Это подтверждает соответствие деятельности компании международным требованиям и открывает перспективы дальнейшего развития для бизнеса.
Стандарты ISO | interstandart
Международная Организации по Стандартизации (ИСО) объединяет национальные организации по стандартизации. Членами ИСО в настоящее время являются более 90 стран. Федеральное агентство по техническому регулированию и метрологии участвует в работе ИСО в качестве национальной организации по стандартизации Российской Федерации.
ФБУ «КВФ «Интерстандарт» заключило лицензионное соглашение с ИСО на право распространения стандартов ИСО на территории РФ, а также на перевод и распространение переводов стандартов ИСО на русском языке.
ФБУ «КВФ «Интерстандарт» предлагает действующие стандарты ISO, поправки к стандартам, отмененные стандарты, проекты и публикации ISO по всем тематикам Международного классификатора стандартов (МКС).
Осуществить поиск и подбор необходимых стандартов ISO Вы можете на официальном сайте организации ИСО: www.iso.org
Стандарты ISO и их переводы распространяются в виде официальных копий, как на бумаге, так и в электронном виде в формате PDF.
Оплата производится по безналичному расчету в рублях на основании выставленного счета.
Стандарты ISO защищены авторским правом. Все права принадлежат организации ISO. Копирование без разрешения правообладателя запрещено!
ПОДРОБНУЮ ИНФОРМАЦИЮ
о стоимости и условиях приобретения стандартов и переводов
Вы можете получить,
направив запрос с перечнем НТД
по e-mail: [email protected]
или по тел.: +7 (499) 236 54 49
Обработка заявки выполняется в течение 2-х рабочих дней
Обновление ценовой информации от 11.01.2021 г.
При изменении курса валюты стоимость на стандарты может быть скорректирована.
Узнать о наличии готового перевода или определить стоимость заказа перевода стандартов на русский язык
Вы можете, направив запрос по e-mail: [email protected]
Международная Организация по стандартизации (ИСО)
Название «ИСО»
Очень много людей замечают несоответствие между полным официальным названием — International Organization for Standardization, и сокращенным — ISO. Получается, что аббревиатурой организации должно быть IOS. Но ISO — это не аббревиатура.
На самом деле, слово ИСО образовано от греческого слова isos, что означает «равный», служит приставкой iso- и встречается в совокупности терминов, таких как «изометрия» (равное измерение или размеры) и «изономия» (равенство закона или людей перед законом).
Название «ИСО» употребляется во всем мире для обозначения организации, во избежание многочисленных вариаций аббревиатуры, получающихся от перевода «International Organization for Standardization» на различные языки членов организаций, например IOS на английском, OIN на французском (от Organisation internationale de normalisation). Таким образом, ИСО — сокращенная форма обозначения международной организации в любой стране.
Таким образом, ИСО — сокращенная форма обозначения международной организации в любой стране.
Как все начиналось
Международная стандартизация начиналась с электротехнической области: Международная электротехническая комиссия (International Electrotechnical Commission — IEC) была создана в 1906 году, а первые работы в других областях начали проводиться такой организацией, как International Federation of the National Standardizing Associations (ISA), учрежденной в 1926 году. Особый акцент в рамках ISA был сделан на машиностроение.
Деятельность ISA прекратилась в 1942 году из-за начала Второй Мировой Войны. Вслед за встречей в Лондоне в 1946 году, делегаты из 25 стран решили создать новую международную организацию «целью которой будет развитие международной координации и объединение промышленных стандартов». Новая организация — ИСО — официально начала функционировать в 23 феврале 1947 года.
Первый стандарт ИСО был выпущен в 1951 году и назывался «Standard reference temperature for industrial length measurement».
Стандарты. Что это такое?
Стандарты — это документированные соглашения, содержащие технически условия или другие точные критерии, которые обычно используются как правила, принципы или определение характеристик для гарантии того, что материалы, продукты, процессы и услуги соответствуют своему назначению.
Например, формат кредитных карт, телефонных карт и «смат» карт разрабатывался в ИСО Международные Стандарты. Соблюдение стандарта, который определяет такой параметр, как оптимальная толщина (0,76 мм), означает, что карты могут использоваться по всему миру.
Таким образом, международные стандарты упрощают жизнь, увеличивают надежность и эффективность товаров и услуг.
Что такое ISO 27001?
Быстрое знакомство с основными фактами
ISO 27001 ― это международный стандарт, разработанный Международной организацией по стандартизации, который описывает, как управлять информационной безопасностью в компании. Последняя версия этого стандарта была опубликована в 2013 году, и его полное название на сегодня ― «ISO/IEC 27001:2013». Первая версия стандарта, опубликованная в 2005 году, базировалась на Британском стандарте BS 7799-2.
Первая версия стандарта, опубликованная в 2005 году, базировалась на Британском стандарте BS 7799-2.
ISO 27001 может быть внедрён в любой организации: коммерческой или некоммерческой, частной или государственной, маленькой или большой. Он был написан ведущими мировыми экспертами в области информационной безопасности и предлагает методологию для внедрения управления информационной безопасностью на предприятии. Он также позволяет компаниям получить сертификацию, что означает, что независимый орган по сертификации подтвердит, что организация внедрила информационную безопасность в соответствии со стандартом ISO 27001.
ISO 27001 стал самым популярным в мире стандартом по информационной безопасности, и многие компании были сертифицированы в соответствии с ним. Здесь Вы можете узнать количество сертификатов за последние два года:
Источник: Исследование Международной организации по стандартизации на тему сертификации по стандарту системы управления
Как работает ISO 27001
Стандарт ISO 27001 сосредоточен на защите конфиденциальности, сохранности и доступности информации в компании. Это реализуется путём выяснения потенциальных проблем с информацией (т.е. оценки рисков), а затем определения необходимых шагов для предотвращения появления таких проблем (т.е. снижения или обработки рисков). Поэтому основная философия ISO 27001 базируется на управлении рисками: выяснить, где находятся риски, а затем систематически обрабатывать их.
Это реализуется путём выяснения потенциальных проблем с информацией (т.е. оценки рисков), а затем определения необходимых шагов для предотвращения появления таких проблем (т.е. снижения или обработки рисков). Поэтому основная философия ISO 27001 базируется на управлении рисками: выяснить, где находятся риски, а затем систематически обрабатывать их.
Защитные меры (или контроли), которые должны внедряться, обычно выступают в форме политик, процедур и технического внедрения (например, программного обеспечения и оборудования). Однако, в большинстве случаев, компании уже располагают у себя всем оборудованием и программным обеспечением. Однако используют они их небезопасным способом, поэтому большая часть внедрений ISO 27001 будет связана с постановкой организационных правил (т.е. с написанием документов), которые необходимы для предотвращения нарушений в системе безопасности. Поскольку такое внедрение потребует управления множеством политик, процедур, людей, активов и т.д., в ISO 27001 описано, как состыковать вместе все эти элементы в системе менеджмента информационной безопасности.
Поэтому управление информационной безопасностью ― это не только ИТ-безопасность (т.е. фаерволы, антивирус и т.д.). Это также управление процессами, правовая защита, управление персоналом, физическая защита и т.д.
См. также: Основная логика ISO 27001: Как работает информационная безопасность?
Почему стандарт ISO 27001 подойдёт Вашей компании?
Вот четыре существенных преимущества в бизнесе, которые может получить компания при внедрении этого стандарта информационной безопасности:
Соответствие правовым требованиям – Появляется всё больше и больше законов, нормативных актов и договорных требований, связанных с информационной безопасностью. И хорошей новостью является то, что многие из них могут быть решены за счёт внедрения ISO 27001, поскольку этот стандарт предоставляет Вам идеальную методологию соблюдения всех их.
Достижение маркетингового преимущества – Если Ваша компания прошла сертификацию, а Ваши конкуренты нет, то Вы можете получить преимущество над конкурентами в глазах Ваших клиентов, которые очень трепетно относятся к вопросу безопасности их информации.
Снижение затрат – Основная философия ISO 27001 ― предотвращать появление инцидентов, связанных с нарушением безопасности, потому что любой инцидент, большой или маленький, стоит денег. Поэтому, предотвращая их, Ваша компания сэкономит довольно много денег. И что самое главное, инвестиции в ISO 27001 гораздо меньше ожидаемой экономии, которую Вы получите.
Улучшение организационного процесса – У типичных быстрорастущих компаний нет времени приостановиться и определить свои процессы и процедуры. Как следствие, сотрудники очень часто просто не знают, что и когда необходимо делать и кем это должно выполняться. Внедрение ISO 27001 помогает решить такие ситуации, потому что это способствует написанию компаниями своих основных процессов (даже тех, которые не связаны с безопасностью) и позволяет сократить потерю времени их сотрудниками.
См. также Бесплатный калькулятор окупаемости инвестиций в безопасность.
Куда вписать управление информационной безопасностью в рамках компании?
Куда вписать управление информационной безопасностью в рамках компании?
В принципе, информационная безопасность является частью управления общими рисками в компании с областями, которые пересекаются с кибербезопасностью, управлением непрерывностью бизнеса и ИТ-управлением:
Как, на самом деле, выглядит ISO 27001?
ISO/IEC 27001 разбит на 11 разделов плюс приложение А. Разделы с 0 по 3 являются вводными (и не обязательны для внедрения), а разделы с 4 по 10 являются обязательными, то есть все их требования должны быть внедрены в организации, если она хочет соответствовать стандарту. Контроли из приложения А должны быть внедрены, только если они заявлены как применимые в Заявлении о применимости.
Разделы с 0 по 3 являются вводными (и не обязательны для внедрения), а разделы с 4 по 10 являются обязательными, то есть все их требования должны быть внедрены в организации, если она хочет соответствовать стандарту. Контроли из приложения А должны быть внедрены, только если они заявлены как применимые в Заявлении о применимости.
Согласно приложению SL Директив ISO/IEC Международной организации по стандартизации, названия разделов в стандарте ISO 27001 аналогичны названиям разделов стандарта ISO 22301:2012, нового стандарта ISO 9001:2015 и других стандартов управления, что позволяет упростить интеграцию этих стандартов.
Раздел 0: Введение – объясняет цель стандарта ISO 27001 и его совместимость с другими стандартами управления.
Раздел 1: Область применения – объясняет, что этот стандарт применим в организации любого типа.
Раздел 2: Нормативные ссылки – относятся к ISO/IEC 27000, как к стандарту, в котором даны термины и определения.
Раздел 3: Термины и определения – опять же относятся к ISO/IEC 27000.
Раздел 4: Особенности организации – Этот раздел является частью фазы планирования в цикле «Планирование, реализация, контроль, корректировка» и определяет требования для понимания внешних и внутренних проблем, заинтересованных сторон и их требований, а также для определения области применения системы менеджмента информационной безопасности.
Раздел 5: Ответственность руководства – Этот раздел является частью фазы планирования в цикле «Планирование, реализация, контроль, корректировка» и определяет обязанности топ-менеджмента, определяет роли и обязанности, а также содержание политики информационной безопасности верхнего уровня.
Раздел 6: Планирование – Этот раздел является частью фазы планирования в цикле «Планирование, реализация, контроль, корректировка» и определяет требования для оценки рисков, обработки рисков, заявления о применимости, плана по обработке рисков и постановки задач для информационной безопасности.
Раздел 7: Поддержка – Этот раздел является частью фазы планирования в цикле «Планирование, реализация, контроль, корректировка» и определяет требования к доступности ресурсов, компетенций, информированности, коммуникации и контролю документации и записей.
Раздел 8: Функционирование – Этот раздел является частью фазы реализации в цикле «Планирование, реализация, контроль, корректировка» и определяет внедрение оценки и обработки рисков, а также контролей и других процессов, необходимых для достижения целей информационной безопасности.
Раздел 9: Оценка эффективности – Этот раздел является частью фазы проверки в цикле «Планирование, реализация, контроль, корректировка» и определяет требования к мониторингу, измерению, анализу, оценке, внутреннему аудиту и анализу управления.
Раздел 10: Усовершенствование – Этот раздел является частью фазы корректировки в цикле «Планирование, реализация, контроль, корректировка» и определяет требования к несоответствиям, исправлениям, корректирующим действиям и непрерывному совершенствованию.
Приложение A – Это приложение содержит каталог из 114 контролей (защитных мер), размещённых в 14 разделах (разделы с A.5 по A.18).
См. также: Был ли в новых версиях стандарта ISO удалён цикл «Планирование, реализация, контроль, корректировка»?
Как внедрить ISO 27001
Чтобы внедрить ISO 27001 в Вашей компании, Вам необходимо выполнить эти 16 шагов:
1) Получить поддержку топ-менеджмента
2) Использовать методологию управления проектом
3) Определить область применения системы менеджмента информационной безопасности
4) Написать политику информационной безопасности верхнего уровня
5) Определить методологию оценки рисков
6) Осуществить оценку и обработку рисков
7) Написать Заявление о применимости
8) Написать план обработки рисков
9) Определить, как измерить эффективность Ваших контролей и Вашей системы менеджмента информационной безопасности
10) Внедрить все соответствующие контроли и процедуры
11) Внедрить программы обучения и информирования
12) Осуществлять всю ежедневную деятельность, прописанную в Вашей документации системы менеджмента информационной безопасности
13) Контролировать и оценивать Вашу систему менеджмента информационной безопасности
14) Осуществить внутренний аудит
15) Осуществить анализ управления
16) Внедрить корректирующие действия
Для более детального объяснения этих шагов ознакомьтесь с перечнем для внедрения ISO 27001.
Обязательная документация
ISO 27001 требует, чтобы была написана следующая документация:
- Область применения системы менеджмента информационной безопасности (пункт 4.3)
- Политика информационной безопасности и задачи (пункты 5.2 и 6.2)
- Методология оценки и обработки рисков (пункт 6.1.2)
- Заявление о применимости (пункт 6.1.3 d)
- План обработки рисков (пункты 6.1.3 e и 6.2)
- Отчёт по оценке рисков (пункт 8.2)
- Определение ролей и обязанностей по обеспечению безопасности (пункты A.7.1.2 и A.13.2.4)
- Инвентаризация активов (пункт A.8.1.1)
- Приемлемое использование активов (пункт A.8.1.3)
- Политика контроля доступа (пункт A.9.1.1)
- Операционные процедуры для ИТ-управления (пункт A.12.1.1)
- Инженерные принципы системы защищённой системы (пункт A.14.2.5)
- Политика безопасности поставщика (пункт A.15.1.1)
- Процедура управления инцидентами (пункт A.
 16.1.5)
16.1.5) - Процедуры непрерывности бизнеса (пункт A.17.1.2)
- Законодательные, нормативные и контрактные требования (пункт A.18.1.1)
А это обязательные записи:
- Записи о подготовке, навыках, опыте и квалификации (пункт 7.2)
- Результаты контроля и измерений (пункт 9.1)
- Программа внутреннего аудита (пункт 9.2)
- Результаты внутренних аудитов (пункт 9.2)
- Результаты анализа управления (пункт 9.3)
- Результаты корректирующих действий (пункт 10.1)
- Журнал действий пользователей, исключений и событий в системе безопасности (пункты A.12.4.1 и A.12.4.3)
Конечно, компания может решить и написать дополнительные документы по безопасности, если сочтёт это необходимым.
Чтобы получить более подробное объяснение каждого из этих документов, скачайте бесплатный перечень обязательной документации, требуемой по ISO 27001:2013.
Как получить сертификацию
Существуют два типа сертификации ISO 27001: (а) для организаций, и (б) для частных лиц. Организации могут пройти сертификацию, чтобы доказать, что они соответствуют всем обязательным пунктам стандарта; частные лица могут посетить курсы и сдать экзамен для того, чтобы пройти сертификацию.
Организации могут пройти сертификацию, чтобы доказать, что они соответствуют всем обязательным пунктам стандарта; частные лица могут посетить курсы и сдать экзамен для того, чтобы пройти сертификацию.
Чтобы получить сертификацию организация должна внедрить стандарт, как это изложено в предыдущих разделах, а затем пройти сертификационный аудит, под руководством органа по сертификации. Сертификационный аудит выполняется в следующей последовательности:
- 1-й этап аудита (обзор документации): аудиторы рассматривают всю документацию.
- 2-й этап аудит (основной аудит): аудиторы проводят аудит на месте, чтобы проверить, совместима ли вся деятельность компании со стандартом ISO 27001 и документацией системы менеджмента информационной безопасности.
- Визиты с инспектированием: после выдачи сертификата в течение 3-летнего его срока действия, аудиторы проверяют, насколько компания поддерживает свою систему менеджмента информационной безопасности.
См. также Прохождение сертификации по ISO 27001 – Как подготовиться к сертификационному аудиту.
также Прохождение сертификации по ISO 27001 – Как подготовиться к сертификационному аудиту.
Частные лица могут пройти несколько курсов для того, чтобы получить сертификаты. Самыми популярными курсами являются:
- Курс «Ведущий аудитор ISO 27001»: этот пятидневный курс обучит Вас, как проводить сертификационный аудит. Он предназначен для аудиторов и консультантов.
- Курс «Ведущий реализатор 27001»: этот пятидневный курс обучит Вас, как внедрять стандарт. Курс предназначен для практиков в области информационной безопасности и для консультантов.
- Курс внутреннего аудитора ISO 27001 ― это двух- или трёхдневный курс, который обучит Вас основам стандарта и как осуществить внутренний аудит. Курс предназначен для новичков в этой теме и для внутренних аудиторов.
См. также: Как узнать о версиях ISO 27001.
Версии 2005 и 2013 стандарта ISO 27001
Как упоминалось ранее, ISO 27001 был впервые опубликован в 2005 году, а его новая версия появилась в 2013 году, поэтому текущая действительная версия ― ISO/IEC 27001:2013.
Самые важные изменения в версии 2013 относятся к структуре основной части стандарта, заинтересованным сторонам, целям, мониторингу и измерению. Кроме того, в приложении A было сокращено количество контролей с 133 до 114 и увеличено количество разделов с 11 до 14. Некоторые требования были удалены из версии 2013, например: предупреждающие действия и требования к документам определённых процедур.
См. также Инфографика: новая версия ISO 27001:2013 – Что изменилось?
Однако, все эти изменения практически не изменили стандарт, в целом. Его основная философия по-прежнему основана на оценке и обработке рисков, а аналогичные фазы в цикле «Планирование, реализация, контроль, корректировка» всё те же. Эту новую версию стандарта проще читать и понимать, и её намного легче интегрировать с другими стандартами управления, такими как ISO 9001, ISO 22301 и т.д.
Компании, которые прошли сертификацию на соответствие требованиям ISO/IEC 27001:2005, должны перейти к новой версии 2013 к сентябрю 2015 года, если они хотят сохранить действительным свой сертификат. Узнайте, как сделать это: How to make a transition from ISO 27001 2005 revision to 2013 revision.
Узнайте, как сделать это: How to make a transition from ISO 27001 2005 revision to 2013 revision.
Связанные стандарты по информационной безопасности и прочие стандарты
Стандарт ISO/IEC 27002 содержит рекомендации по внедрению контролей, указанных в ISO 27001. Стандарт ISO 27001 указывает на 114 контролей, которые можно использовать, чтобы снизить риски для безопасности. ISO 27002 может быть весьма полезен, потому что он содержит подробности того, как внедрить эти контроли. Стандарт ISO 27002, именуемый ранее как ISO/IEC 17799, появился на основе Британского стандарта BS 7799-1.
ISO/IEC 27004 содержит рекомендации для оценки информационной безопасности. Этот стандарт хорошо сочетается с ISO 27001, потому что объясняет, как определить, достигла ли система менеджмента информационной безопасности своих целей.
ISO/IEC 27005 содержит рекомендации для управления рисками нарушения информационной безопасности. Это очень хорошее дополнение к стандарту ISO 27001, потому что содержит детали того, как осуществлять оценку и обработку рисков (а это, возможно, самый сложный этап внедрения). ISO 27005 появился на основе Британского стандарта BS 7799-3.
ISO 22301 определяет требования к системам управления непрерывностью бизнеса. Стандарт очень хорошо сочетается с ISO 27001, потому что A.17 стандарта ISO 27001 требует внедрения непрерывности бизнеса, но не предоставляет слишком много подробностей. Узнайте больше о ISO 22301 здесь…
ISO 9001 определяет требования к системам менеджмента качества. И хотя, на первый взгляд, у менеджмента качества и менеджмента информационной безопасности не так много общего, но, на самом деле, порядка 25% требований стандартов ISO 27001 и ISO 9001 аналогичны: управление документацией, внутренний аудит, анализ управления, корректирующие действия, постановка задач и управление компетенциями. Это значит, что если компания внедрила ISO 9001, то внедрить ISO 27001 будет намного проще. Узнайте больше о ISO 9001 здесь…
Секвенирование
полноразмерных транскриптов (Iso-Seq) — CD Genomics
CD Genomics имеет большой опыт в предоставлении услуг Iso-Seq путем создания полноразмерных транскриптов без сборки. После каждой процедуры проводится строгий контроль качества, чтобы гарантировать полные и точные результаты.
Введение Iso-Seq
У эукариотических организмов один ген может кодировать удивительное количество белков после альтернативного сплайсинга, каждый из которых выполняет свою биологическую функцию.Известны гены человека, которые выполняют очень разные функции в зависимости от того, какой вариант сплайсинга экспрессируется. С альтернативным сплайсингом, столь важным для функционирования генома.
Short-read RNA-Seq работает путем физического разделения изоформ транскриптов на более мелкие части и повторной сборки, оставляя возможности для неправильной сборки или неполного захвата всего разнообразия изоформ из интересующих генов. Важно записывать полные стенограммы. Используя преимущества технологии секвенирования длинных считываний PacBio SMRT, секвенирование изоформ (Iso-Seq) может легко покрыть полный транскрипт от 5′-конца до 3′-поли A-хвоста без необходимости фрагментации для получения полноразмерных последовательностей кДНК, который полезен для идентификации новых транскриптов и новых интронов, таким образом точно идентифицируя изоформы, альтернативные сайты сплайсинга, экспрессию слитых генов и аллельную экспрессию.
По сравнению с платформой Illumina, анализ PacBio может легко обнаружить очень длинные полицистронные молекулы РНК (называемые сложными транскриптами) и выявить широкий спектр новых транскрипционных перекрытий между соседними и удаленными генами, расположенными параллельно. Полезно повысить возможность изучения общегеномной сети, осуществляющей совместный контроль над экспрессией и репликацией генов.
Рабочий процесс проекта
Требования к образцам:
- Количество РНК: Общая РНК ≥ 5 мкг (без деградации или загрязнения ДНК)
- : OD260 / 280 = 1.8 ~ 2.2; OD260 / 230 ≥ 1,5
- Качество РНК: 28S: 18S ≥ 1,5 , RIN ≥ 7
Чистота РНК
Стратегия секвенирования:
Платформы PacBio, тип библиотеки> 4K, 6 ~ 10 Гбайт / образец
Анализ данных
Транскриптом без ссылки
Стандартный анализ данных
- Полноразмерный анализ изоформы, включая коррекцию изоформы полной длины, классификацию, уменьшенную избыточность
- , включая генную онтологию, путь KEGG, KOG или COG, Swissport
- Анализ генных структур, включая альтернативный сплайсинг, LncRNA, SSR, CDS
Аннотации транскриптома
Расширенный анализ данных
- Карта для ссылки
- Анализ уровня экспрессии генов
- Анализ дифференциально экспрессируемых генов
- Обогащенный анализ дифференциально экспрессируемого транскриптома KEGG
- Тепловая карта дифференциально экспрессируемого гена
- … (подробнее по запросу)
Транскриптом со ссылкой
Стандартный анализ данных
- Полноразмерный анализ изоформы, включая картирование эталонного генома, полноразмерную коррекцию изоформы, классификацию, уменьшенную избыточность
- , включая генную онтологию, путь KEGG, KOG или COG, Swissport
- Анализ генных структур, включая альтернативный сплайсинг, LncRNA, SSR, CDS, предсказание нового транскриптома, идентификация гибридного гена
Аннотации транскриптома
Расширенный анализ данных
- Карта для ссылки
- Анализ уровня экспрессии генов
- Анализ дифференциально экспрессируемых генов
- Анализ обогащения дифференциально экспрессируемого транскриптома KEGG.
- Тепловая карта дифференциально экспрессируемого гена
- … (подробнее по запросу)
Аналитический трубопровод
Длинное секвенирование позволяет напрямую идентифицировать альтернативно транскрибируемые или процессируемые транскрипты, полицистронные единицы транскрипции и другие длинные последовательности кДНК. Благодаря опыту и целеустремленности передовая платформа PacBio SMRT и услуги от CD Genomics станут вашим лучшим спутником в Iso-Seq. Пожалуйста, свяжитесь с нами для получения дополнительной информации и подробного предложения.
* Только для исследовательских целей. Не использовать в диагностических процедурах.
Утилита PacBio Iso-Seq для обнаружения транскриптов и генов в латексе гевеи
An D, Cao H, Li C, Humbeck K, Wang W (2018) Секвенирование изоформ и современные приложения для разгадки сложности транскриптомы растений. Гены 9 (1): 43
Google Scholar
Berthelot K, Lecomte S, Estevez Y, Peruch F (2014) Hevea brasiliensis REF (Hev b 1) и SRPP (Hev b 3): обзор белков резиновых частиц.Биохимия 106: 1–9
CAS
Google Scholar
Bokma E, Barends T, Terwissch van Scheltingab AC, Dijkstr BW, Beintema JJ (2000) Ферментная кинетика гевамина, хитиназы из каучукового дерева Hevea brasiliensis . FEBS Lett 478: 119–122
CAS
Google Scholar
Bokma E, Rozeboom HJ, Sibbald M, Dijkstra BW, Beintema JJ (2002) Экспрессия и характеристика мутантов активного сайта гевамина, хитиназы из каучукового дерева Hevea brasiliensis .Eur J Biochem 269: 893–901
CAS
Google Scholar
Bokma E, Spiering M, Chow KS, Mulder PP, Subroto T, Beintema JJ (2001) Определение последовательностей кДНК и геномной ДНК гевамина, хитиназы из каучукового дерева Hevea brasiliensis . Plant Physiol Biochem 39: 367–376
CAS
Google Scholar
Bokma E, van Koningsveld GA, Jeronimus-Stratingh M, Beintema JJ (1997) Гевамин, хитиназа из каучукового дерева Hevea brasiliensis , расщепляет пептидогликан между C-1 из N -ацетилглюкозы. C-4 из N -ацетилмурамовой кислоты и, следовательно, не является лизоцимом.FEBS Lett 411 (2–3): 161–163
CAS
Google Scholar
Камачо К., Кулурис Г., Авагян В., Ма Н., Пападопулос Дж., Билер К., Мэдден Т.Л. (2009) BLAST +: архитектура и приложения. BMC Bioinform 10: 421
Google Scholar
Chao J, Chen Y, Wu S, Tian WM (2015) Сравнительный анализ транскриптома латекса из клона каучукового дерева CATAS8-79 и PR107 обнаруживает новые сигналы для регуляции регенерации латекса и продолжительности потока латекса.BMC Plant Biol 15 (1): 104
Google Scholar
Cheng B, Furtado A, Henry RJ (2017) Длительное секвенирование транскриптома кофейных зерен выявляет разнообразие полноразмерных транскриптов. Gigascience 6 (11): gix86
Google Scholar
Чоу К.С., Ван К.Л., Мат Иса М.Н., Бахари А., Тан С.Х., Харикришна К., Йанг Х.Й. (2007) Анализ биосинтеза каучука на основе транскриптомного анализа латекса Hevea brasiliensis .J Exp Bot 58: 2429–2440
CAS
Google Scholar
Чоу К.С., Мат Иса М.Н., Бахари А., Газали А.К., Псевдоним Х., Мохд-Зайнуддин З., Хо С.К., Ван К.Л. (2012) Метаболические пути, влияющие на биосинтез каучука в латексе Hevea brasiliensis . J Exp Bot 63: 1863–1871
CAS
Google Scholar
Chow KS, Ghazali AK, Hoh CC, Mohd-Zainuddin Z (2014) Требование глубины чтения секвенирования РНК для оптимального охвата транскриптомом в Hevea brasiliensis .BMC Res Notes 7:69
Google Scholar
Conesa A, Gotz S, Garcia-Gomez JM, Terol J, Talon M, Robles M (2005) Blast2GO: универсальный инструмент для аннотации, визуализации и анализа в исследованиях функциональной геномики. Биоинформатика 21: 3674–3676
CAS
Google Scholar
Dong L, Liu H, Zhang J, Yang S, Kong G, Chu JSC, Chen N, Wang D (2015) Одномолекулярное секвенирование транскриптов в реальном времени облегчает аннотацию генома мягкой пшеницы и исследование транскриптома зерна.BMC Genom 16: 1039
Google Scholar
Duan C, Argout X, Gébelin V, Summo M, Dufayard JF, Leclercq J, Piyatrakul P, Pirrello J, Rio M, Champion A, Montoro P (2013) Идентификация суперсемейства Hevea brasiliensis AP2 / ERF путем секвенирования РНК. BMC Genom 14 (1): 30
CAS
Google Scholar
Филичкин С.А., Гамильтон М., Дхармавардхана П.Д., Сингх С.К., Салливан С., Бен-Гур А., Редди А.С., Джайсвал П. (2018) Абиотические стрессы модулируют ландшафт транскриптома тополя посредством альтернативного сплайсинга, дифференциального удержания интронов и переключение соотношения изоформ.Фронтальный завод Sci 12 (9): 5
Google Scholar
Gordon SP, Tseng E, Salamov A, Zhang J, Meng X, Zhao Z, Kang D, Underwood J, Grigoriev IV, Figueroa M, Schilling JS, Chen F, Wang Z (2015) Широко распространенные полицистронные расшифровки у грибов, выявленных с помощью секвенирования мРНК одной молекулы. PLoS One 10: e0132628
Google Scholar
Goyvaerts E, Dennis M, Light D., Chua NH (1991) Клонирование и секвенирование кДНК, кодирующей фактор удлинения каучука Hevea brasiliensis .Физиология растений 97 (1): 317–321
CAS
Google Scholar
Hoang NV, Furtado A, Mason PJ, Marquardt A, Kasirajan L, Thirugnanasambandam PP, Botha FC, Henry RJ (2017) Обзор сложного транскриптома из высокополиплоидного генома сахарного тростника с использованием полноразмерного изоформного секвенирования и сборка de novo из короткой последовательности чтения. BMC Genom 18 (1): 395
Google Scholar
Huelsenbeck JP, Ronquist F (2001) MRBAYES: Байесовский вывод филогенетических деревьев. Биоинформатика 17 (8): 754–755
CAS
Google Scholar
Jekel PA, Hartmann BH, Beintema JJ (1991) Первичная структура гевамина, фермента с лизоцим-хитиназной активностью из латекса Hevea brasiliensis . Eur J Biochem 200: 123–130
CAS
Google Scholar
Jiao WB, Schneeberger K (2017) Влияние геномных технологий третьего поколения на сборку генома растений. Curr Opin Plant Biol 36: 64–70
CAS
Google Scholar
Jo IH, Lee J, Hong C, Lee D, Bae W, Park SG, Ahn Y, Kim Y, Kim J, Lee J, Hyun D (2017) Секвенирование изоформ дает более полное представление о panax ginseng транскриптом. Гены 8 (9): 228
Google Scholar
Ko JH, Chow KS, Han KH (2003) Анализ транскриптома показывает новые особенности молекулярных событий, происходящих в латициферах Hevea brasiliensis (пара каучуковое дерево). Plant Mol Biol 53: 479–492
CAS
Google Scholar
Лау Н.С., Макита Ю., Кавашима М., Тейлор Т.Д., Кондо С., Осман А.С., Чонг ASC, Мацуи М. (2016) Геном каучукового дерева показывает расширение семейства генов, связанного с биосинтезом каучука.Научный представитель 6: 28594
CAS
Google Scholar
Li C, Lin F, An D, Wang W, Huang R (2017) Секвенирование и сборка генома с помощью длинных считываний в растениях. Гены 9 (1): 6
Google Scholar
Li Y, Wei W, Feng J, Luo H, Pi M, Liu Z, Kang C (2017) Повторная аннотация генома дикой земляники Fragaria vesca с использованием обширной РНК на основе Illumina и SMRT -seq наборы данных.ДНК Res 25 (1): 61–70
Google Scholar
Liu JP, Xia ZQ, Tian XY, Li YJ (2015) Секвенирование транскриптома и анализ каучукового дерева ( Hevea brasiliensis Muell.) Для обнаружения предполагаемых генов, связанных с сухостью панелей (TPD). BMC Genom 16 (1): 398
Google Scholar
Lopez D, Amira MB, Brown D, Muries B, Brunel-Michac N, Bourgerie S, Porcheron B, Lemoine R, Chrestin H, Mollison E, Di Cola A, Frigerio L, Julien JL, Gousset- Dupont A, Fumanal B, Label P, Pujade-Renaud V, Auguin D, Venisse JS (2016) Подсемейство аквапоринов Hevea brasiliensis XIP: геномные, структурные и функциональные характеристики, имеющие отношение к интенсивному сбору латекса.Plant Mol Biol 91: 375–396
CAS
Google Scholar
Mantello CC, Cardoso-Silva CB, Da Silva CC, De Souza LM, Scaloppi EJ Jr, de Goncalves PS, Vicentini R, De Souza AP (2014) Сборка De novo и анализ транскриптома каучукового дерева ( Hevea brasiliensis ) и разработка маркеров SNP для путей биосинтеза каучука. PLoS One 9: e102664
Google Scholar
Montoro P, Wu S, Favreau B, Herlinawati E, Labrune C, Martin-Magniette ML, Pointet S, Rio M, Leclercq J, Ismawanto S (2018) Анализ транскриптома в латексе Hevea brasiliensis выявил изменения в сигнальных путях гормонов во время Этефонная стимуляция и последующее высыхание постукивающей панели. Научный доклад 8 (1): 8483
Google Scholar
Oh SK, Kang H, Shin DH, Yang J, Chow KS, Yeang HY, Wagner B, Breiteneder H, Han KH (1999) Выделение, характеристика и функциональный анализ нового клона кДНК, кодирующего небольшой белок частиц каучука из Hevea brasiliensis .J Biol Chem 274 (24): 17132–17138
CAS
Google Scholar
Pertea M, Kim D, Pertea GM, Leek JT, Salzberg SL (2016) Анализ экспрессии на уровне транскрипта в экспериментах с последовательностью РНК с HISAT, StringTie и Ballgown. Nat Protoc 11: 1650–1667
CAS
Google Scholar
Pootakham W, Sonthirod C, Naktang C, Ruang-Areerate P, Yoocha T, Sangsrakru D, Theerawattanasuk K, Rattanawong R, Lekawipat N, Tangphatsornruang S (2017) показывает гибридную сборку гена каучукового дерева De novo свидетельства палеотетраплоидии у гевей видов.Научный представитель 7: 41457
CAS
Google Scholar
Робертс Р.Дж., Карнейро М.С., Шатц М.С. (2013) Преимущества секвенирования SMRT. Геном Биол 14 (6): 405
Google Scholar
Робертс А., Трапнелл С., Донаги Дж., Ринн Дж. Л., Пахтер Л. (2011) Улучшение оценок экспрессии RNA-Seq путем корректировки смещения фрагментов. Геном Биол 12: R22
CAS
Google Scholar
Робинсон Дж. Т., Торвальдсдоттир Х., Винклер В., Гуттман М., Ландер Э. С., Гетц Г., Месиров Дж. П. (2011) Программа для просмотра интегративной геномики. Nat Biotechnol 29 (1): 24
CAS
Google Scholar
Ronquist F, Teslenko M, Van Der Mark P, Ayres DL, Darling A, Höhna S, Larget B, Liu L, Suchard MA, Huelsenbeck JP (2012) MrBayes 3.2: эффективный байесовский филогенетический вывод и выбор модели в большом пространстве модели. Syst Biol 61 (3): 539–542
Google Scholar
Rozeboom HJ, Budiani A, Beintema JJ, Dijkstra BW (1990) Кристаллизация гевамина, фермента с лизоцим / хитиназной активностью из латекса Hevea brasiliensis . J Mol Biol 212: 441–443
CAS
Google Scholar
Salgado LR, Koop DM, Pinheiro DG, Rivallan R, Le Guen V, Nicolás MF, De Almeida LG, Rocha VR, Magalhães M, Gerber AL, Figueira A (2014) De novo анализ транскриптома Hevea brasiliensis с помощью РНК-секвенирования и скрининга молекулярных маркеров.BMC Genom 15 (1): 236
Google Scholar
Schulz MH, Zerbino DR, Vingron M, Birney E (2012) Oases: надежная сборка de novo последовательности РНК в динамическом диапазоне уровней экспрессии. Биоинформатика 28: 1086–1092
CAS
Google Scholar
Sievers F, Wilm A, Dineen D, Gibson TJ, Karplus K, Li W, Lopez R, McWilliam H, Remmert M, Söding J, Thompson JD (2011) Быстрое масштабируемое производство высококачественного белка множественное выравнивание последовательностей с использованием Clustal Omega.Мол Сист Биол 7 (1): 539
Google Scholar
Симау Ф.А., Уотерхаус Р.М., Иоаннидис П., Кривенцева Е.В., Здобавов Е.М. (2015) БУСКО: оценка сборки генома и полноты аннотации с помощью ортологов с единственной копией. Биоинформатика 31: 3210–3212
Google Scholar
Subroto T, van Koningsveld GA, Schreuder HA, Soedjanaatmadja UM, Beintema JJ (1996) Хитиназа и β-1,3-глюканаза во фракции лютоидных тел в латексе Hevea .Фитохимия 43 (1): 29–37
CAS
Google Scholar
Tan D, Hu X, Fu L, Kumpeangkeaw A, Ding Z, Sun X, Zhang J (2017) Сравнительный анализ морфологии и транскриптома показывает различные функции первичных и вторичных клеток латицифера в каучуковом дереве. Научный доклад 7 (1): 3126
Google Scholar
Tang C, Xiao X, Li H, Fan Y, Yang J, Qi J, Li H (2013) Сравнительный анализ латексного транскриптома показывает предполагаемые молекулярные механизмы, лежащие в основе сверхпродуктивности Hevea brasiliensis .PLoS One 8: e75307
CAS
Google Scholar
Тан Ц, Ян М, Фанг Y, Ло И, Гао С, Сяо X, Ан З, Чжоу Б, Чжан Б, Тан Х, Йанг Х., Цинь И, Ян Дж, Линь Q, Мэй Х , Montoro P, Long X, Qi J, Hua Y, He Z, Sun M, Li W, Zeng X, Cheng H, Liu Y, Yang J, Tian W, Zhuang N, Zeng R, Li D, He P, Li Z, Zou Z, Li S, Li C, Wang J, Wei D, Lai CQ, Luo W, Yu J, Hu S, Huang H (2016) Геном каучукового дерева позволяет по-новому взглянуть на производство каучука и адаптацию видов.Nat Plants 2: 16073
CAS
Google Scholar
Terwisscha van Scheltinga AC, Hennig M, Dijkstra BW (1996) Структура разрешения 1,8 A гевамина, растительной хитиназы / лизоцима и анализ консервативных последовательностей и структурных мотивов семейства гликозилгидролаз 18. J Mol Биол 262: 243–257
CAS
Google Scholar
Terwisscha van Scheltinga AC, Kalk KH, Beintema JJ, Dijkstra BW (1994) Кристаллические структуры гевамина, защитного белка растений с хитиназной и лизоцимной активностью и его комплекса с ингибитором.Структура 2: 1181–1189
CAS
Google Scholar
Thorvaldsdóttir H, Robinson JT, Mesirov JP (2013) Integrative Genomics Viewer (IGV): высокопроизводительная визуализация и исследование данных геномики. Краткий Биоинформ 14 (2): 178–192
Google Scholar
Trapnell C, Hendrickson DG, Sauvageau M, Goff L, Rinn JL, Pachter L (2013) Дифференциальный анализ регуляции генов при разрешении транскриптов с помощью RNA-seq.Nat Biotechnol 31: 46–53
CAS
Google Scholar
Tungngoen K, Viboonjun U, Kongsawadworakul P, Katsuhara M, Julien JL, Sakr S, Chrestin H, Narangajavana J (2011) Гормональная обработка коры каучуковых деревьев ( Hevea brasiliensis –30) увеличивает урожайность латексов. разведение латекса в связи с дифференциальной экспрессией двух генов аквапоринов. J Plant Physiol 168 (3): 253–262
CAS
Google Scholar
Wang B, Tseng E, Regulski M, Clark TA, Hon T, Jiao Y, Lu Z, Olson A, Stein JC, Ware D (2016) Раскрытие сложности транскриптома кукурузы с помощью одномолекулярного долгого чтения секвенирования. Nat Commun 7: 11708
CAS
Google Scholar
Wang M, Wang P, Liang F, Ye Z, Li J, Shen C, Pei L, Wang F, Hu J, Tu L, Lindsey K (2018) Глобальный обзор альтернативного сращивания аллополиплоидного хлопка : ландшафт, сложность и регуляция.Новый Фитол 217 (1): 163–178
Google Scholar
Wu TD, Watanabe CK (2005) GMAP: программа геномного картирования и выравнивания последовательностей мРНК и EST. Биоинформатика 21: 1859–1875
CAS
Google Scholar
Xia Z, Xu H, Zhai J, Li D, Luo H, He C, Huang X (2011) Анализ РНК-Seq и сборка транскриптома de novo Hevea brasiliensis .Завод Мол Биол 77: 299–308
CAS
Google Scholar
Yuan Y, Bayer PE, Batley J, Edwards D (2017) Улучшения в геномных технологиях: применение в геномике сельскохозяйственных культур. Trends Biotechnol 35 (6): 547–558
CAS
Google Scholar
Zerbino DR (2010) Использование ассемблера velvet de novo для технологий короткого чтения. Curr Protoc Bioinform 31 (1): 11.5.1–11.5.12
Google Scholar
Чжан В., Циклитира П., Мессинг Дж. (2013) Секвенирование семейств генов с помощью PacBio — тематическое исследование с генами глютена пшеницы. Ген 533: 541–546
Google Scholar
Zhang G, Sun M, Wang J, Lei M, Li C, Zhao D, Huang J, Li W, Li S, Li J, Yang J (2019) Полноразмерное секвенирование кДНК PacBio, интегрированное с РНК -seq reads значительно улучшает обнаружение транскриптов сплайсинга в рисе.Завод J 97 (2): 296–305
CAS
Google Scholar
Zhu C, Li X, Zheng J (2018) Профилирование транскриптома с использованием последовательности РНК острого перца на основе Illumina и SMRT для глубокого понимания генов, участвующих в инфекции CMV. Ген 666: 123–133
CAS
Google Scholar
Zou Z, Gong J, An F, Xie G, Wang J, Mo Y, Yang L (2015) Полногеномная идентификация каучукового дерева ( Hevea brasiliensis Muell.Arg.) Гены аквапоринов и их реакция на стимуляцию этефоном в латицифере, ткани, продуцирующей каучук. BMC Genom 16: 1001
Google Scholar
Zuo C, Blow M, Sreedasyam A, Kuo RC, Ramamoorthy GK, Torres-Jerez I, Li G, Wang M, Dilworth D, Barry K, Udvardi M (2018) Раскрытие транскриптомической сложности просо просо Последовательность длинных считываний PacBio. Биотехнология Биотопливо 11 (1): 170
Google Scholar
Страница не найдена — PacBio
Соглашение об использовании изображения
Загружая, копируя или используя изображения, размещенные на этом веб-сайте («Сайт»), вы подтверждаете, что прочитали, поняли и согласны с условиями настоящего Соглашения об использовании изображений, а также с условиями, приведенными на веб-страницу Юридические уведомления, которые вместе регулируют использование вами изображений, как указано ниже.Если вы не согласны с такими условиями, не загружайте, не копируйте и не используйте изображения каким-либо образом, если у вас нет письменного разрешения, подписанного уполномоченным представителем Pacific Biosciences.
В соответствии с условиями настоящего Соглашения и условиями, приведенными на веб-странице Юридических уведомлений (в той степени, в которой они не противоречат условиям настоящего Соглашения), вы можете использовать изображения на Сайте исключительно для (а) редакционного использования в прессе. и / или отраслевых аналитиков, (б) в связи с обычной, рецензируемой, научной публикацией, книгой или презентацией и т.п.Вы не можете изменять или модифицировать любое изображение, полностью или частично, по любой причине. Вы не можете использовать какое-либо изображение, искажающее представление о связанных продуктах, услугах или технологиях Pacific Biosciences или о любых связанных характеристиках, данных или их свойствах. Вы также не можете использовать любое изображение способом, который обозначает какое-либо представление или гарантию (явную, подразумеваемую или установленную законом) от Pacific Biosciences в отношении продукта, услуги или технологии. Права, предоставляемые настоящим Соглашением, являются личными для вас и не могут быть переданы вами другой стороне.
Вы, а не Pacific Biosciences, несете ответственность за использование изображений. Вы признаете и соглашаетесь с тем, что любое неправильное использование изображений или нарушение настоящего Соглашения нанесет компании Pacific Biosciences непоправимый вред. Pacific Biosciences является владельцем или лицензиатом изображения, но не агентом владельца. Вы соглашаетесь предоставить Pacific Biosciences следующую кредитную линию: «Предоставлено Pacific Biosciences of California, Inc., Menlo Park, CA, USA», а также включаете любые другие кредиты или благодарности, отмеченные Pacific Biosciences.Вы должны включить любое уведомление об авторских правах, изначально включенное в изображения, на всех копиях.
ИЗОБРАЖЕНИЙ ПРЕДОСТАВЛЯЮТСЯ Pacific Biosciences «КАК ЕСТЬ». Pacific Biosciences ОТКАЗЫВАЕТСЯ ОТ ВСЕХ ЗАЯВЛЕНИЙ И ГАРАНТИЙ, ЯВНЫХ, ПОДРАЗУМЕВАЕМЫХ ИЛИ ЗАКОНОДАТЕЛЬНЫХ УСЛОВИЙ, ВКЛЮЧАЯ, НО НЕ ОГРАНИЧИВАЯСЬ НАРУШЕНИЕМ, СОБСТВЕННОСТЬЮ, КОММЕРЧЕСКОЙ ЦЕННОСТЬЮ И ПРИГОДНОСТЬЮ ДЛЯ ОПРЕДЕЛЕННОЙ ЦЕЛИ. НИ ПРИ КАКИХ ОБСТОЯТЕЛЬСТВАХ Pacific Biosciences НЕ НЕСЕТ ОТВЕТСТВЕННОСТИ ЗА ЛЮБЫЕ ПРЯМЫЕ, КОСВЕННЫЕ, СЛУЧАЙНЫЕ, КАЧЕСТВЕННЫЕ ИЛИ КОСВЕННЫЕ УБЫТКИ ЛЮБОГО ТИПА, КАСАЮЩИЕСЯ ИЗОБРАЖЕНИЙ.
Вы соглашаетесь с тем, что Pacific Biosciences может прекратить ваш доступ и использование изображений, размещенных на веб-сайте PacificBiosciences.com, в любое время и без предварительного уведомления, если компания сочтет, что вы нарушили какое-либо из условий настоящего Соглашения об использовании изображений. Вы соглашаетесь возмещать, защищать и оградить Pacific Biosciences, ее должностных лиц, директоров, сотрудников, агентов, лицензиаров, поставщиков и любых сторонних поставщиков информации на Сайт от всех убытков, расходов, убытков и издержек, включая разумные гонорары адвокатам. в результате любого нарушения вами условий настоящего Соглашения об использовании изображений или прекращения Pacific Biosciences вашего доступа к Сайту или его использования.Прекращение действия не повлияет на права Pacific Biosciences или ваши обязательства, возникшие до прекращения.
Страница не найдена — PacBio
Соглашение об использовании изображения
Загружая, копируя или используя изображения, размещенные на этом веб-сайте («Сайт»), вы подтверждаете, что прочитали, поняли и согласны с условиями настоящего Соглашения об использовании изображений, а также с условиями, приведенными на веб-страницу Юридические уведомления, которые вместе регулируют использование вами изображений, как указано ниже.Если вы не согласны с такими условиями, не загружайте, не копируйте и не используйте изображения каким-либо образом, если у вас нет письменного разрешения, подписанного уполномоченным представителем Pacific Biosciences.
В соответствии с условиями настоящего Соглашения и условиями, приведенными на веб-странице Юридических уведомлений (в той степени, в которой они не противоречат условиям настоящего Соглашения), вы можете использовать изображения на Сайте исключительно для (а) редакционного использования в прессе. и / или отраслевых аналитиков, (б) в связи с обычной, рецензируемой, научной публикацией, книгой или презентацией и т.п.Вы не можете изменять или модифицировать любое изображение, полностью или частично, по любой причине. Вы не можете использовать какое-либо изображение, искажающее представление о связанных продуктах, услугах или технологиях Pacific Biosciences или о любых связанных характеристиках, данных или их свойствах. Вы также не можете использовать любое изображение способом, который обозначает какое-либо представление или гарантию (явную, подразумеваемую или установленную законом) от Pacific Biosciences в отношении продукта, услуги или технологии. Права, предоставляемые настоящим Соглашением, являются личными для вас и не могут быть переданы вами другой стороне.
Вы, а не Pacific Biosciences, несете ответственность за использование изображений. Вы признаете и соглашаетесь с тем, что любое неправильное использование изображений или нарушение настоящего Соглашения нанесет компании Pacific Biosciences непоправимый вред. Pacific Biosciences является владельцем или лицензиатом изображения, но не агентом владельца. Вы соглашаетесь предоставить Pacific Biosciences следующую кредитную линию: «Предоставлено Pacific Biosciences of California, Inc., Menlo Park, CA, USA», а также включаете любые другие кредиты или благодарности, отмеченные Pacific Biosciences.Вы должны включить любое уведомление об авторских правах, изначально включенное в изображения, на всех копиях.
ИЗОБРАЖЕНИЙ ПРЕДОСТАВЛЯЮТСЯ Pacific Biosciences «КАК ЕСТЬ». Pacific Biosciences ОТКАЗЫВАЕТСЯ ОТ ВСЕХ ЗАЯВЛЕНИЙ И ГАРАНТИЙ, ЯВНЫХ, ПОДРАЗУМЕВАЕМЫХ ИЛИ ЗАКОНОДАТЕЛЬНЫХ УСЛОВИЙ, ВКЛЮЧАЯ, НО НЕ ОГРАНИЧИВАЯСЬ НАРУШЕНИЕМ, СОБСТВЕННОСТЬЮ, КОММЕРЧЕСКОЙ ЦЕННОСТЬЮ И ПРИГОДНОСТЬЮ ДЛЯ ОПРЕДЕЛЕННОЙ ЦЕЛИ. НИ ПРИ КАКИХ ОБСТОЯТЕЛЬСТВАХ Pacific Biosciences НЕ НЕСЕТ ОТВЕТСТВЕННОСТИ ЗА ЛЮБЫЕ ПРЯМЫЕ, КОСВЕННЫЕ, СЛУЧАЙНЫЕ, КАЧЕСТВЕННЫЕ ИЛИ КОСВЕННЫЕ УБЫТКИ ЛЮБОГО ТИПА, КАСАЮЩИЕСЯ ИЗОБРАЖЕНИЙ.
Вы соглашаетесь с тем, что Pacific Biosciences может прекратить ваш доступ и использование изображений, размещенных на веб-сайте PacificBiosciences.com, в любое время и без предварительного уведомления, если компания сочтет, что вы нарушили какое-либо из условий настоящего Соглашения об использовании изображений. Вы соглашаетесь возмещать, защищать и оградить Pacific Biosciences, ее должностных лиц, директоров, сотрудников, агентов, лицензиаров, поставщиков и любых сторонних поставщиков информации на Сайт от всех убытков, расходов, убытков и издержек, включая разумные гонорары адвокатам. в результате любого нарушения вами условий настоящего Соглашения об использовании изображений или прекращения Pacific Biosciences вашего доступа к Сайту или его использования.Прекращение действия не повлияет на права Pacific Biosciences или ваши обязательства, возникшие до прекращения.
Введение в метод Iso-Seq: секвенирование полноразмерных транскриптов
из блога PacBio
С недавним запуском SMRT Analysis v2.2, мы рады представить программное обеспечение для анализа, поддерживающее новый метод Iso-Seq ™ для секвенирования полноразмерных транскриптов и изоформ генов без необходимости сборки! Сегодня мы более подробно рассмотрим метод Iso-Seq, чтобы объяснить его уникальную научную ценность, и рассмотрим публикации тех, кто уже применяет секвенирование одиночных молекул в реальном времени (SMRT®) в этой захватывающей области исследований.
В геномах растений и животных, наряду со всеми высшими эукариотическими организмами, большинство генов подвергается альтернативному сплайсингу с образованием множественных изоформ транскриптов. У людей, например, есть данные об альтернативном сплайсинге более 95% генов [1], в среднем более пяти изоформ на ген. Регулирование генов посредством альтернативного сплайсинга может резко увеличить потенциал кодирования белков генома, который содержит ограниченное количество генов, кодирующих белки.Несколько удивительно, что альтернативно сплайсированные изоформы одного гена также могут иметь очень разные, даже антагонистические, функции [2]. Следовательно, понимание функциональной биологии генома требует знания полного набора изоформ. Микромассивы и высокопроизводительное секвенирование кДНК стали невероятно полезными инструментами для изучения транскриптомов, однако эти технологии предоставляют небольшие фрагменты транскриптов, и создание полных транскриптов для изучения изоформ генов было сложной задачей.
Благодаря необычайно длинным считываниям, доступным при секвенировании PacBio®, новый метод Iso-Seq обеспечивает считывание полной длины, охватывающее полные изоформы транскрипта от полиА-хвоста до 5 ‘конца.Больше нет необходимости реконструировать транскрипты или делать выводы изоформ на основе объединения локальной информации, поскольку каждая последовательность представляет собой отдельную полноразмерную молекулу кДНК. Этот метод сочетает в себе разрешение на уровне изоформ с лучшим секвенированием всего транскриптома, чтобы обеспечить прямое секвенирование изоформ гена по всему транскриптому. Мы рады сообщить, что ученые теперь используют метод Iso-Seq для рутинной секвенирования полноразмерных изоформ у самых разных организмов и применяют этот подход для улучшения аннотаций в эталонных геномах, характеристики изоформ генов в важных семействах генов и находить новые гены даже в наиболее изученных линиях клеток человека.
Вот избранный список последних публикаций, презентаций и примеров данных:
• Au et al. (2013) Характеристика транскриптома ESC человека с помощью гибридного секвенирования. PNAS 110: E4821-4830.
• Sharon et al. (2013) Одномолекулярный долгосрочный обзор человеческого транскриптома. Nature Biotechnol 31: 1009-1014.
• Thomas et al. (2014) Долгосрочное секвенирование куриных транскриптов и идентификация новых изоформ транскриптов. PLoS One.9: e94650.
• Treutlein et al. (2014) Картография альтернативного сплайсинга нейрексина, картированная с помощью секвенирования мРНК с длительным считыванием одной молекулы. PNAS 111: E1291-1299.
• Zhang et al. (2014) PacBio-секвенирование семейств генов — тематическое исследование с генами глютена пшеницы. Gen 533: 541-546.
• Бринзевич Д. и др. (2014) ВИЧ-1 взаимодействует с оболочками эндогенного ретровируса К человека (HML-2), полученными из первичных лимфоцитов человека. Журнал вирусологии 88: 6213-6223.
• П.Ларсен и др. (2012) Применение циклического консенсусного секвенирования и сетевого анализа для характеристики репертуара бычьего IgG. BMC Immunology, 13: 52
• Веб-семинар: Сборка не требуется — очень длинные чтения для полноразмерного секвенирования изоформ транскрипта
• Набор данных Iso-Seq MCF-7 человека
Для получения более подробной информации о секвенировании кДНК с помощью PacBio не пропустите этот праймер на GitHub и общий протокол на SampleNet.
[1] Пан и др. (2008) Глубокое исследование альтернативной сложности сплайсинга в человеческом транскриптоме с помощью высокопроизводительного секвенирования.Природа Генетики 40: 1413-1415.
[2] Boise et al. (1993) Bcl-x, ген, родственный bcl-2, который функционирует как доминантный регулятор апоптотической гибели клеток. Ячейка 74: 597-608.
границ | Анализ транскриптома и эпитранскриптома в растениях с использованием PacBio Iso-Seq и прямого секвенирования РНК на основе нанопор
Введение
Анализ транскриптомов, которые представляют активность генов в геноме, жизненно важен для понимания взаимосвязи между генотипом и фенотипом.Динамика и сложность транскриптома регулируют все аспекты роста, развития растений и реакции на различные внешние биотические и абиотические сигналы. Различные методы, такие как секвенирование экспрессирующей метки последовательности (EST) (Wu et al., 2002), серийный анализ экспрессии генов (SAGE) (Matsumura et al., 1999), ДНК-микрочип (Hihara et al., 2001) и недавно Секвенирование РНК (RNA-Seq) с использованием технологий секвенирования следующего поколения (NGS) (Mortazavi et al., 2008) было разработано для анализа транскриптомов.С 2005 года платформы короткого чтения второго поколения быстро вытеснили технологию секвенирования Sanger первого поколения для различных высокопроизводительных приложений из-за более низких затрат и большей глубины секвенирования (Sedlazeck et al., 2018). Тем не менее, длина считывания является основным ограничением в секвенировании короткого считывания второго поколения, что затрудняет анализ некоторых аспектов событий совместной / посттранскрипционной обработки. Чтобы преодолеть это ограничение, в последние несколько лет исследователи секвенируют полноразмерные транскрипты в основном с использованием двух платформ: Pacific BioSciences (PacBio) (Rhoads and Au, 2015) и Oxford Nanopore Technologies (ONT) (Bayega et al., 2018), которые называются технологиями секвенирования «третьего» и «четвертого» поколения соответственно (Slatko et al., 2018). Эти две платформы значительно увеличили длину чтения по сравнению с другими методами NGS и, следовательно, могут использоваться для решения более широкого круга исследовательских вопросов. Одномолекулярное секвенирование изоформ в реальном времени (SMRT) (Iso-Seq) с использованием платформы PacBio захватывает полную длину транскриптов (Gonzalez-Garay, 2016) и, таким образом, представляет более простые и точные способы для различных приложений, таких как аннотация генов (Zhao и другие., 2018), идентификация изоформ (Abdel-Ghany et al., 2016; Wang T. et al., 2017), идентификация гибридных транскриптов (Weirather et al., 2015) и открытие длинных некодирующих РНК (lncRNA) ( Ли и др., 2016). Здесь мы обсуждаем приложения и более широкое применение PacBio и ONT в исследованиях транскриптомов. Недавно разработанная прямая RNA-Seq с использованием нанопор позволяет избежать ошибок амплификации (Garalde et al., 2018). Кроме того, эта технология может предоставить полное представление о модификациях РНК, таких как N 6 -метиладенозин, 5-метилцитидин и 5-гидроксилметилцитидин (Li X.et al., 2017), которые вместе именуются «эпитранскриптомом».
Части основного алгоритма для анализа длинного чтения PacBio и ONT аналогичны стратегиям анализа короткого чтения, используемым в подходах к секвенированию второго поколения. Тем не менее, для некоторых приложений были разработаны специальные новые инструменты биоинформатики, которые не были частью конвейеров секвенирования второго поколения. Эти инструменты необходимы для обеспечения большей гибкости для достижения различных целей, а также для решения новых проблем, таких как более высокая частота ошибок и низкая пропускная способность.Мы представляем доступные в настоящее время биоинформатические методы анализа считывания PacBio и ONT, включая извлечение интересующих считываний (ROI), исправление ошибок (Au et al., 2012), отображение (Wu and Watanabe, 2005), кластеризацию изоформ (Fu et al. ., 2012), а также идентификацию изоформ множественных транскриптов (Abdel-Ghany et al., 2016). Улучшения в этих новых методах и вычислительных конвейерах расширят ландшафт сложности транскриптома на уровне изоформы транскрипта и эпитранскриптома с более высокой пропускной способностью и более высокой точностью.Здесь мы обсудили методологии PacBio Iso-Seq и ONT direct RNA-Seq, текущее состояние инструментов биоинформатики, используемых для анализа длинных считываний, и выделили различные применения этих методов.
Подготовка библиотеки и извлечение считываемой вставки из Pacbio Iso-Seq
Как правило, высококачественная РНК представляет собой поли (A), выбранный для создания библиотек последовательного секвенирования PacBio с использованием, например, набора Clontech SMARTer PCR (Ramsköld et al., 2012; Li et al., 2016). Длина считывания секвенирования зависит от качества РНК и генерации полноразмерных кДНК.Для обогащения полноразмерных кДНК в библиотеке был использован метод лигирования кэп-зависимого линкера (Cartolano et al., 2016). Альтернативно полноразмерные РНК могут быть обогащены путем комбинирования отбора РНК поли (A) + с захватом 5′-кэпированных мРНК с использованием кэп-связывающего белка (Blower et al., 2013). Затем полноразмерная мРНК используется для синтеза первой цепи кДНК с олиго (dT) праймером с последующим синтезом второй цепи кДНК с выбором размера полноразмерной кДНК в нескольких различных размерах (Xu et al., 2015). С новой системой Sequel кДНК можно секвенировать без выбора размера. Путем лигирования адаптеров шпильки к двухцепочечной кДНК создаются библиотеки SMRTbell TM , которые впоследствии можно секвенировать на платформе RSII или Sequel (Xu et al., 2017). Сравнение 5′-концов с аннотированными стартовыми сайтами транскриптов показало, что этот протокол обеспечивает полноразмерное секвенирование кДНК с небольшой потерей 5′- или 3′-концов (Ramsköld et al., 2012).
В настоящее время PacBio предлагает два секвенсора четвертого поколения: RSII был первым коммерчески доступным инструментом для секвенирования, а недавно улучшенное устройство Sequel обеспечивает гораздо более высокую пропускную способность (до 20 Гб на ячейку SMRT).Стратегия секвенирования PacBio основана на использовании технологии волноводов с нулевой модой (ZMW), которая состоит из крошечных нано-лунок, первоначально описанных в 2003 году (Levene et al., 2003). ZMWs позволяют иммобилизовать шаблоны секвенирования посредством взаимодействия с механизмом секвенирования, комплексом полимеразных ферментов, который прикреплен к основанию ZMWs (Rhoads and Au, 2015). Затем включение флуоресцентно меченых оснований ДНК испускает флуоресцентные сигналы, которые улавливаются детектором в реальном времени (McCarthy, 2010).Адаптеры шпильки, которые добавляются к обоим концам двухцепочечной ДНК во время подготовки библиотеки, создают замкнутую кольцевую матрицу ДНК, которую можно многократно пересекать полимеразой с длительным сроком службы для повышения точности. Таким образом, платформа PacBio может генерировать несколько подпотоков, включая последовательности адаптеров, в одном ZMW и давать непрерывное долгое чтение (CLR), которое может генерировать более точные чтения циклической консенсусной последовательности (CCS) (Weirather et al., 2017).
Впоследствии система RSII и система Sequel сохраняют данные основного вызова и соответствующие показатели качества в формате файлов HDF5 и BAM соответственно.Инструмент bax2bam может конвертировать формат файла HDF5 в формат BAM.
Модуль анализа SMRT от SMRT Link от PacBio адаптирован для получения эффективных подпотоков (рисунок 1). Затем извлечение ROI для каждого ZMW является вторым шагом в рабочем процессе биоинформатического анализа PacBio Iso-Seq. Этот шаг выполняется с конвейером SMRT Link, который включает в себя шаги по обрезке адаптеров и генерации CCS. Затем области интереса очищаются от поли-A / T-хвостов, праймеров, искусственных конкатемеров и определяется направление цепи транскрипта (Bayega et al., 2018). Конвейер ToFu Pacbio из пакета SMRT Analysis можно использовать для поиска адаптеров секвенирования для извлечения ROI и полноразмерных нехимерных (FLNC) чтений (Wang T. et al., 2017; Xu et al., 2017). После этого считывания FLNC, которые содержат как 5 ‘, так и 3’ праймеры и поли-A-хвост, могут быть проанализированы с использованием итеративной кластеризации для исправления ошибок (ICE) для построения консенсусных кластеров для повышения согласованной точности. Впоследствии PacBio RS II и Sequel используют Quiver и Arrow для доработки консенсусных последовательностей соответственно (Bayega et al., 2018).
Рисунок 1. Различные приложения и биоинформатические решения для прямого секвенирования РНК PacBio Iso-Seq и Nanopore в растениях. Iso-seq и прямое секвенирование РНК могут быть выполнены с использованием анализа SMRT от PacBio и MinKNOW от ONT, соответственно. Затем эти длинные считывания в формате FASTA будут проходить этап исправления ошибок перед последующим анализом. Чтобы иметь дело с различными приложениями (средние поля), вычислительные инструменты, используемые для обработки и анализа длинных считываний для каждого приложения, указаны для видов без и со справочными геномами в левом и правом прямоугольниках соответственно.
Создание библиотеки и вызов оснований для прямого секвенирования РНК в нанопорах
Стартовый пакет для прямого RNA-Seq стоит всего 1000 долларов США (цены по состоянию на январь 2019 г.) и включает в себя один секвенатор MinION, две проточные кюветы, один набор для секвенирования и набор для промывки. По сравнению с NGS или PacBio, MinION — портативное (весит 90 г), устройство с длительным считыванием в реальном времени и недорогое устройство. Также возможно использовать протокол SMARTer для синтеза полноразмерной кДНК (Ramsköld et al., 2012), который включает в себя репарацию концов, dA-хвост и лигирование адаптера.Однако такая конструкция библиотеки удалит всю информацию о модификации РНК во время синтеза двухцепочечной кДНК. Рабочий процесс создания библиотеки прямой РНК Nanopore использует адаптер poly (dT) и обратную транскриптазу SuperScript III для создания гибридов РНК-ДНК, которые впоследствии лигируются с адаптерами секвенирования нанопор с использованием ДНК-лигазы Т4 перед секвенированием. Затем магнитные шарики Agencourt RNAClean XP используются для очистки дуплексов РНК-ДНК. После оценки концентрации образца библиотека прямой РНК Nanopore может быть загружена в проточные кюветы с помощью секвенатора MinION, GridION или PromethION.По сравнению с MinION секвенсоры GridION и PromethION обеспечивают более высокую пропускную способность. Моторный белок тянет 3′-конец цепи РНК внутрь канала нанопоры (Bayega et al., 2018). Затем датчик регистрирует изменения ионного тока в каждой поре.
Перед секвенированием компьютерное оборудование должно быть проверено на соответствие минимальным требованиям. Минимальные требования для MinION: ЦП: i7 или Xeon с 4+ ядрами, память: 16 ГБ ОЗУ, хранилище: внутренний SSD 1 ТБ, порты: USB3.На любом компьютере с указанными выше минимальными требованиями можно запустить MinION без снижения производительности во время секвенирования. Настольный или портативный компьютер с установленным агентом MinKNOW и EPI2ME Desktop Agent, предоставленным Oxford Nanopore и Metrichor Ltd. соответственно, необходимо подключить к MinION (рис. 1). MinKNOW управляет устройством MinION, тестирует оборудование, проверяет проточные кюветы и выполняет запуски секвенирования. EPI2ME дополнительно анализирует генерируемые необработанные электрические сигналы и сохраняет их в файлах FAST5, которые служат входными данными для Metrichor при вызове базы.Затем последовательности FASTQ и FASTA могут быть извлечены из файлов FAST5 с помощью инструментов poretools (Loman and Quinlan, 2014). Однако средства обнаружения модификаций базы в настоящее время недоступны. Большинство приложений, использующих Nanopore direct RNA-Seq, были сосредоточены на млекопитающих. В настоящее время Nanopore direct RNA-Seq не сообщалось об исследованиях на растениях. Однако ожидается, что все больше и больше лабораторий будут использовать этот инструмент для изучения модификаций РНК в растениях.
Предварительная обработка длинного чтения: исправление ошибок, сопоставление и кластеризация длинных чтений
Хотя длина чтения PacBio и ONT больше, чем у NGS, одна общая проблема, связанная с этими технологиями, — высокая частота ошибок (Koren et al., 2012). Таким образом, перед последующим использованием необходимо уменьшить количество ошибок. В настоящее время исправление считываний PacBio и ONT делится на три отдельные категории: гибридная стратегия исправления ошибок, метод самокоррекции и исправление ошибок на основе эталонных данных.
Гибридная стратегия исправления ошибок использует короткие чтения из NGS для исправления длинных считываний. LSC (Au et al., 2012), LoRDEC (Salmela and Rivals, 2014) и PacBioToCA (Koren et al., 2012) — три широко используемых метода исправления ошибок (рисунок 1).В отличие от LSC и PacBioToCA, LoRDEC избегает сопоставления коротких чтений за счет построения графа Де Брюйна коротких чтений (DBG) порядка k и распределяет длинные чтения через эту DBG коротких чтений для исправления. Таким образом, LoRDEC требует меньше времени / памяти и меньше места на диске (Salmela and Rivals, 2014). Недавно Nanocorr был разработан специально для исправления длинных считываний Nanopore с использованием высококачественных коротких считываний (Goodwin et al., 2015).
В качестве альтернативы, программное обеспечение самокоррекции отличается от вышеупомянутой гибридной стратегии исправления ошибок, которая зависит от коротких чтений.Long-read multiple aligner (LoRMA) — один из методов исправления ошибок, основанный только на длинных чтениях (Salmela et al., 2016). По сравнению с другим методом самокоррекции, алгоритмом PacBio corrected reads (PBcR) (Koren et al., 2012), LoRMA обеспечивает более высокую пропускную способность и меньшую частоту ошибок. Однако для получения точной коррекции метод самокоррекции требует большого охвата, что ограничивает его применение.
Третий метод обеспечивает исправление ошибок на основе ссылок во время сопоставления длинных считываний с эталонным геномом, и некоторые инструменты, которые делают этот тип исправления ошибок, — это minimap2 (Li, 2018) и minialign.Это быстрые и точные инструменты выравнивания для длинных считываний PacBio и Nanopore с высокой частотой ошибок при вставке и удалении. Конвейер анализа транскриптомов от Isoform Sequencing (TAPIS) (Abdel-Ghany et al., 2016) также выполняет исправление ошибок на основе справочных данных. В дополнение к minimap2, GMAP (Wu and Watanabe, 2005) и STAR (Dobin et al., 2013) представляют собой два выравнивателя с поддержкой сплайсинга, которые можно использовать для сопоставления полноразмерных считываний с эталонным геномом для последующего анализа. Однако GMAP и STAR не выполняют исправление ошибок во время сопоставления.В дополнение к каноническим сайтам сплайсинга GMAP и STAR захватывают неканонические сайты сплайсинга, поэтому следует проявлять осторожность при последующем анализе AS. По сравнению с GMAP, minimap2 более соответствует существующим аннотациям и хорошо работает с зашумленным чтением (Li, 2018).
Высокоэкспрессируемые гены могут генерировать несколько идентичных изоформ, что потребует больше времени для последующей обработки / анализа и их трудно визуализировать без сокращения избыточных считываний. На этапе кластеризации можно сгруппировать полноразмерные считывания в кластер, что является необходимым шагом для дальнейшего повышения качества и выявления уникальных изоформ сплайсинга.После сопоставления Iso-Seq с эталонным геномом Cupcake ToFU можно использовать для уничтожения избыточных изоформ и получения уникальных изоформ. Большинство стратегий кластеризации, используемых для видов без эталонного генома, были разработаны для EST, которые появились до эпохи PacBio и ONT. Программы кластеризации, разработанные для EST, такие как UCLUST (Edgar, 2010) и CD-HIT (Fu et al., 2012), широко используются для группировки и уничтожения избыточных последовательностей. Однако эти методы не были разработаны для полноразмерных последовательностей с высоким уровнем ошибок по сравнению с EST или короткими чтениями из NGS.В настоящее время существует два алгоритма de novo для кластеризации длинных чтений по генам: алгоритм ICE (Gordon et al., 2015) может кластеризовать чтения FLNC из секвенирования PacBio для генерации консенсусных изоформ и алгоритм CARNAC-LR, разработанный для ONT. данные долгочитаемого секвенирования (Marchet et al., 2018). После свертывания избыточных изоформ информация о счетчике чтения для уровней экспрессии будет потеряна. Если необходимо выполнить анализ уровня экспрессии, можно вернуться и получить счетчики чтения из исходных файлов секвенирования.
Приложения и инструменты биоинформатики для прямого секвенирования РНК Iso-Seq и Nanopore в растениях
В настоящее время глубокое секвенирование PacBio и ONT все чаще используется для аннотации генома, идентификации ко / посттранскрипционных событий и транскриптов слияния. Недавно в нескольких исследованиях были собраны и повторно проанализированы длинные считывания из Iso-Seq в комплексные базы данных, такие как база данных секвенирования Plant ISOform (PISO) (Feng et al., 2019) и ISOdb (Xie et al., 2018). ISOdb и PISO депонировали 8 и 19 видов соответственно.Поскольку новая технология имеет более высокое разрешение, чем секвенирование второго поколения, и обнаруживает модифицированные основания РНК, дополнительные аспекты регуляции транскрипции и посттранскрипции могут быть изучены более всесторонне. Поэтому мы выделяем биоинформатические решения и различные приложения, которые сложно исследовать с помощью NGS.
De Novo Аннотации генома, идентификация нового локуса и коррекция генной модели
Для видов без доступного эталонного генома, таких как Drynaria roosii (Sun et al., 2018) и Asparagus officinalis (Kakrana et al., 2018), Iso-Seq недавно был успешно использован для захвата полного и полноразмерного транскриптома. Из-за более длинных считываний из PacBio и ONT, Iso-Seq оказался более эффективным в разрешении многих сложных особенностей транскриптомов по сравнению с коротким считыванием RNA-Seq, которое зависит от программного обеспечения для восстановления последовательностей транскриптов (Haas et al., 2013; Steijger et al., 2013). Таким образом, одним из ключевых преимуществ длинных считываний из PacBio и ONT было точное определение генных моделей путем генерации полноразмерных транскриптов без дальнейшей сборки, что является сложной задачей для сложных изоформ (Gordon et al., 2015). О полезности длинночитываемых транскриптов в моделях предполагаемых генов сообщалось в лекарственной траве Panax ginseng (Jo et al., 2017; Kim et al., 2018), аллогексаплоидной пшенице (Clavijo et al., 2017), мягкой пшенице. (Cartolano et al., 2016), сахарная свекла (Minoche et al., 2015), кофейные зерна (Cheng et al., 2017) и каучуковое дерево Para (Pootakham et al., 2017). Полноразмерные транскрипты, генерируемые Iso-Seq, идеально подходят для улучшения предсказания генной модели и идентификации новых генов, которые не отображаются на аннотированные генные локусы.Например, недавние исследования выявили 2171 новый ген у Sorghum bicolor (Abdel-Ghany et al., 2016), 8091 у Phyllostachys edulis (Wang T. et al., 2017) и 3026 у Triticum aestivum ( Гордон и др., 2015). Также в Populus trichocarpa (Filichkin et al., 2018), аллополиплоидный хлопок (Wang et al., 2018) и Populus « Nanlin 895 » (Chao Q. et al., 2018), 15 087, 13 551 , и 1575 новых транскрибируемых областей, соответственно, были недавно идентифицированы.В дополнение к идентификации изоформы и нового локуса, Iso-Seq был использован для уточнения генных моделей в Vitis vinifera cv. Каберне Совиньон (Minio et al., 2018) и аллополиплоидный хлопок (Wang et al., 2018). Кроме того, недавние исследования исправили 178 и 2241 аннотированных генов, которые охватывали более одной сборки транскриптов в S. bicolor (Abdel-Ghany et al., 2016) и P. edulis (Wang T. et al., 2017). , соответственно. Программа для сборки сплайсированных выравниваний (PASA) — это один из инструментов биоинформатики, который исправляет такие аннотации генов (Haas et al., 2008). Недавно конвейер длинных аннотаций (LoReAn) использовал комбинацию длинных чтений PacBio SMRT или MinION и другую информацию, такую как данные о белках для аннотации генов (Cook et al., 2019).
Характеристика альтернативной инициации транскрипции, альтернативного полиаденилирования и альтернативного сплайсинга
Альтернативная инициация транскрипции (ATI), альтернативное расщепление и альтернативное полиаденилирование (APA) и альтернативный сплайсинг (AS) являются тремя основными процессами, которые вносят вклад в разнообразие транскриптомов.AS мРНК-предшественников (пре-мРНК) может потенциально увеличить количество белковых изоформ, продуцируемых мультиэксонными генами, и регулировать экспрессию генов с помощью множества механизмов, таких как измененная эффективность трансляции изоформ сплайсинга, распад, не опосредованный смыслом, и деградация мРНК под воздействием лекарств. (Редди и др., 2013). Хотя отдельные события AS могут быть количественно оценены и аннотированы с помощью NGS с большой точностью, трудно вывести изоформы сплайсинга полной длины, которые содержат комбинацию этих отдельных событий AS (Steijger et al., 2013). Секвенирование с длительным считыванием обеспечивает возможность получения полноразмерных последовательностей и, таким образом, идентификации сложных изоформ сплайсинга, которые трудно обнаружить и реконструировать с помощью NGS. Iso-Seq позволил идентифицировать более 110 000 неизбыточных изоформ в Zea mays (Wang et al., 2016),> 42 000 в P. edulis (Wang T. et al., 2017) и> 16000 из Salvia miltiorrhiza (Xu et al., 2015). Кроме того, Iso-Seq идентифицировал 29730 новых изоформ у Trifolium pratense L., 2501 новый альтернативный транскрипт в V. vinifera cv. Каберне Совиньон (Minio et al., 2018) и более 11000 новых изоформ сплайсинга у S. bicolor L. Moench (Abdel-Ghany et al., 2016). Для 35,74% видов бермудских трав с помощью Iso-Seq были идентифицированы три или более различных изоформ (Zhang B. et al., 2018). В дикой землянике Fragaria vesca Iso-Seq обнаружил, что пре-мРНК из ~ 58% мультиэксонных генов альтернативно сплайсируются (Li Y. et al., 2017).
В дополнение к детекции полноразмерных изоформ, события AS можно разделить на пять различных типов: сохраненный интрон (RI), пропущенный экзон (SE), альтернативный 5′-сайт сплайсинга (A5SS), альтернативный 3′-сайт сплайсинга (A3SS) , и взаимоисключающие экзоны (Shen et al., 2014). Помимо перечисленных выше пяти общих категорий, существует множество других сложных типов, таких как альтернативное положение, то есть альтернативные 3 ‘и 5’ сайты (Wang and Brendel, 2006), AS и инициация транскрипции (ASTI) (Nagasaki et al., 2006) альтернативные первые экзоны (Chen et al., 2007) и составные паттерны (Wang and Rio, 2018). Хотя NGS может обнаруживать эти события AS, длинные чтения из PacBio и ONT обеспечивают преимущество при обнаружении событий AS, поскольку секвенирование длительного чтения может избежать любых возможных проблем во время реконструкции транскриптома. Например, Iso-Seq выявил 10053, 172 743, 133 229 и 21 154 события AS у S. bicolor (Abdel-Ghany et al., 2016), Z. mays (Wang et al., 2016), аллополиплоидного хлопка. (Ван и др., 2018) и P. edulis (Wang T. et al., 2017) соответственно.
Альтернативное полиаденилирование выполняет множество регуляторных ролей в транспортировке, локализации, стабильности и трансляции РНК, производя изоформы с разными сайтами 3′-расщепления, что создает разнообразие и сложность транскриптов (Tilgner et al., 2015; Abdel-Ghany et al., 2016; Wang T. et al., 2017). Для идентификации APA с использованием NGS библиотеки Poly (A) Site Sequencing (PAS-Seq) могут быть сконструированы с использованием вырожденных нуклеотидов в комбинации с олиго (T) праймерами (Shepard et al., 2011; Zhang et al., 2015). Проблема внутреннего праймирования была определена как гибридизация праймеров кДНК с внутренним непрерывным As вместо фактического поли (A) хвоста (Beaudoing et al., 2000). Если в окне 10 нт присутствовало шесть непрерывных As или более семи As, это был кандидат на внутреннее праймирование (Tian et al., 2005). PAS-Seq на основе методов NGS не может избежать внутреннего прайминга, потому что внутренние A-богатые последовательности могут праймировать олиго (dT) (Nam et al., 2002; Sherstnev et al., 2012). Оба метода Iso-Seq и Nanopore direct RNA-Seq позволяют избежать внутреннего прайминга.Используя Iso-Seq, недавно было обнаружено 7700 генов, содержащих два или более сайта полиаденилирования в S. bicolor (Abdel-Ghany et al., 2016). У аллополиплоидного хлопка 6935 генов имеют как минимум пять поли (A) сайтов (Wang et al., 2018). В настоящее время количественный анализ APA все еще зависит от NGS из-за низкой глубины последовательности Iso-Seq и Nanopore direct RNA-Seq. В недавнем исследовании P. edulis использовался метод, сочетающий NGS с Iso-Seq для идентификации 1224 дифференциальных сайтов APA (Wang T.и др., 2017). Ожидается, что в будущем как Iso-Seq, так и Nanopore direct RNA-Seq можно будет использовать для количественного анализа после увеличения пропускной способности.
Альтернативная инициация транскрипции — еще один ключевой механизм генерации разнообразных транскриптов (Tanaka et al., 2009). Альтернативное использование сайтов старта транскрипции привлекло мало внимания у растений по сравнению с исследованиями AS и APA. Стратегия парного анализа стартовых сайтов транскрипции (PEAT), которая требует сложной конструкции библиотеки, после NGS использовалась для мониторинга глобального использования сайтов начала транскрипции (Ni et al., 2010). Используя протокол PEAT, в корнях Arabidopsis были идентифицированы миллионы сайтов начала транскрипции, которые попадают в три категории (Morton et al., 2014). Поскольку PacBio Iso-Seq и Nanopore direct RNA-Seq могут секвенировать полноразмерные транскрипты от 5′-концов до полиаденилированных хвостов, это было бы идеальным инструментом для обнаружения ATI.
Для традиционной RNA-Seq идентификация основных событий AS, включая события пропуска экзонов, удержание интронов, использование альтернативного 5′-донора и альтернативного 3′-донора, довольно просто с помощью нескольких инструментов, включая rMATS (Shen et al., 2014), JUM (Wang and Rio, 2018), PASA pipeline (Campbell et al., 2006) и ASTALAVISTA (Foissac and Sammeth, 2007). Для анализа посттранскрипционной регуляции, основанной на секвенировании с длинным считыванием, TAPIS pipeline (Abdel-Ghany et al., 2016) и PRAPI (Gao et al., 2017) являются двумя основными инструментами биоинформатики, которые используют Iso-Seq для идентификации AS и APA (рисунок 1). Кроме того, PRAPI (Gao et al., 2017) может также идентифицировать несколько других событий / процессов, таких как ATI и производство кольцевых РНК (circRNAs).
Идентификация транскриптов слияния
Fusion транскриптов являются результатом события транс-сплайсинга (Li et al., 2008), которое объединяет две отдельно кодируемые пре-РНК в один транскрипт. Транскрипты слияния были идентифицированы у различных видов растений (Zhang et al., 2010; Wang et al., 2016). Наборы данных парных концов RNA-Seq, основанные на NGS, были успешно проанализированы на предмет транскрипта слияния (Maher et al., 2009). Недавно Iso-Seq предоставил более надежный способ идентификации транскриптов слияния.Всего с помощью Iso-Seq было обнаружено 1430 слитых транскриптов у Z. mays (Wang et al., 2016). Кроме того, 3762 и 222 слитых транскрипта были идентифицированы у T. pratense L (Chao Y. et al., 2018) и аллополиплоидного хлопка (Wang et al., 2018) соответственно.
Стандарт для идентификации транскриптов слияния основан на простой идее, что два или более фрагмента из одного транскрипта могут быть картированы в несколько локусов (Wang et al., 2016). Были разработаны несколько алгоритмов обнаружения транскриптов слияния на основе NGS (Liu S.и др., 2015). Однако эти алгоритмы были специально разработаны для данных с парным концом RNA-Seq. Пакет PacBio pbtranscript-ToFU предоставляет сценарий для обнаружения транскриптов слияния (fusion_finder.py), который специально разработан для чтения из Iso-Seq. Обнаружение и прогнозирование изоформ (IDP) слияние (рис. 1) также представляет другой алгоритм для обнаружения событий слияния с использованием как длинного секвенирования PacBio, так и NGS (Weirather et al., 2015).
Идентификация днРНК
Длинные нкРНК определяются как РНК, содержащие более 200 нуклеотидов и не имеющие заметного кодирующего потенциала (Jin et al., 2013). У растений днРНК могут генерироваться из межгенных, интронных или кодирующих областей и играть важную роль в регуляции генов (Wang and Chekanova, 2017). Большинство днРНК в растениях полиаденилировано, поэтому RNA-Seq на платформах Illumina также может определять экспрессию днРНК. Однако недавние исследования показали, что lncRNAs подвергаются сложной посттранскрипционной регуляции (Liu J. et al., 2015). Таким образом, полноразмерное секвенирование дает большое преимущество в идентификации генной модели днРНК.Недавно в нескольких исследованиях сообщалось об идентификации днРНК с использованием Iso-Seq в растениях. Например, PacBio Iso-Seq выявил 1187 и 4333 днРНК у тополя « Nanlin 895 » (Chao Q. et al., 2018) и T. pratense L. (Chao Y. et al., 2018), соответственно. . Эти исследования показали, что Iso-Seq хорошо подходит для идентификации днРНК. GreeeNC и CANTATAdb — два ресурса для поиска гомологии последовательностей днРНК из длинных считываний, о которых сообщалось в P.edulis (Wang T. et al., 2017). Кроме того, длинные чтения, содержащие гомологию последовательностей с miRNA, также могут рассматриваться как некодирующие РНК, как сообщалось в S. bicolor (Abdel-Ghany et al., 2016). В Z. mays lncRNAs были идентифицированы с использованием PLEK, модели классификации, обученной на известных lncRNAs с высокой степенью достоверности (Wang et al., 2016).
Идентификация природных антисмысловых транскриптов
Естественные антисмысловые транскрипты (NAT), включая типы «голова к голове», «хвост к хвосту» и полностью перекрывающиеся типы, как было показано, действуют в регуляции транскрипции и посттранскрипции генов (Faghihi and Wahlestedt, 2009).Всего 932 цис -NAT были идентифицированы с использованием набора данных PacBio SMRT, специфичного для цепи, путем попарного сравнения перекрывающихся координат из противоположно ориентированных полноразмерных транскриптов (Zhang H. et al., 2018). Кроме того, PRAPI был разработан для идентификации NAT на основе длинных чтений PacBio / ONT (Gao et al., 2017). В то же время PRAPI может также количественно определять экспрессию NAT, комбинируя чтения NGS с использованием конструкции библиотеки, специфичной для цепи (рисунок 1).
Анализ длинных считываний в отсутствие эталонного генома
В связи с недавними разработками в области секвенирования с длинным считыванием, все больше и больше исследований по секвенированию генома используют платформы с длинным считыванием для получения более длинных считываний, чем N50, например, de novo сборка травы Oropetium thomaeum (VanBuren et al., 2015), подсолнечника (Badouin et al., 2017) и цитрусовых (Wang X. et al., 2017). Однако есть еще много видов, у которых нет доступных последовательностей генома. Таким образом, будет полезно разработать безреференсный анализ аннотации транскрипции с использованием Trinity (Haas et al., 2013) и других инструментов для посттранскрипционного анализа. Недавние исследования показали, что возможно реконструировать модели полноразмерных транскриптов для видов без эталонного генома, таких как Astragalusmbranaceus (Li J.et al., 2017), Arabidopsis pumila (Yang et al., 2018) и Zanthoxylum bungeanum Maxim (Tian et al., 2018) с использованием длинных чтений.
Недавно в AS_ de _ novo было сообщено об идентификации AS на основе Iso-Seq без эталонных геномов (Liu et al., 2017). Основная идея возникла из поиска делеции или вставки в единицах кластеризации (Ner-Gaon et al., 2007; Zhou et al., 2011; Wu et al., 2014; Liu et al., 2017). Таким образом, кластеризация длинных чтений из PacBio Iso-Seq или ONT должна быть первым шагом перед идентификацией AS.Для этого анализа можно использовать несколько программ кластеризации, например широко используемую CD-HIT (Fu et al., 2012). Недавно был выпущен один подход к кластеризации, разработанный для длинных чтений Oxford Nanopore (Marchet et al., 2018). После этапа кластеризации сравнение BLAT все против всех может использоваться для идентификации сегментации вставки или удаления, вызванной событиями AS (Liu et al., 2017). Гибридное секвенирование и поиск карты (HySeMaFi) объединили PacBio Iso-Seq и NGS для выявления сплайсинга и количественной оценки изоформ (Ning et al., 2017). AStrap адаптировал модель машинного обучения для идентификации событий AS путем интеграции более 500 встроенных функций (Ji et al., 2018).
Применение прямого секвенирования РНК с помощью нанопор
Поскольку полноразмерная нативная РНК-Seq (nRNA-Seq) ONT обеспечивает множество преимуществ по сравнению с NGS, этот метод был применен для обнаружения вирусных транскриптомов (Moldován et al., 2018a), модификаций оснований 16S рРНК (Smith et al. , 2017), вирусного патогена (Depledge et al., 2018) и идентификации изоформ искусственного сплайсинга во время обратной транскрипции из-за переключения матрицы (Moldován et al., 2018б). Наконец, значительным преимуществом прямого RNA-Seq является то, что он позволяет обнаруживать ко / посттранскрипционные модификации оснований в РНК, поскольку не требует этапов обратной транскрипции и ПЦР-амплификации. Многие обратимые химические модификации оснований происходят в мРНК, которые в совокупности называются «эпитранскриптомом» (Gilbert et al., 2016). Эти ковалентные обратимые химические модификации нуклеотидов регулируют многие аспекты экспрессии генов. Недавние исследования показывают, что эпитранскриптомные модификации играют ключевую роль в регуляции сплайсинга пре-мРНК, ядерного экспорта, стабильности и локализации мРНК, а также эффективности трансляции (Gilbert et al., 2016; Xiao et al., 2016; Roundtree et al., 2017; Slobodin et al., 2017), а также несколько процессов развития у растений (Fray, Simpson, 2015; Vandivier, Gregory, 2018). Не существует простого высокопроизводительного инструмента для обнаружения модификаций мРНК и их динамики у растений. Широко используемый метод анализа модификаций РНК в масштабе всего транскриптома является сложной задачей, поскольку для каждой модификации требуются специфические антитела. Эти антитела затем используются для осаждения РНК с модификациями, которая затем подвергается высокопроизводительному секвенированию (рис. 2).Этот метод был использован для идентификации m 6 A по всему транскриптому и численности у животных (Dominissini et al., 2012; Meyer et al., 2012). В Arabidopsis thaliana сообщалось о профилях 6-метиладенина (m 6 A) и 5-метилцитозина (m 5 C) по всему транскриптому с использованием m 6 A- или m 5 C-нацеленных. антитела, соответственно, для иммунопреципитации РНК (RIP) с последующим высокопроизводительным секвенированием (m 6 A-Seq / m 5 C-Seq) (Luo et al., 2014; Cui et al., 2017). Этот подход RIP-Seq имеет несколько ограничений, включая потребность в специфических антителах для каждой модификации. Это также требует много времени и усилий. Кроме того, трудно получить достаточное количество иммунопреципитированной РНК. Что еще более важно, этот метод не обеспечивает точное местоположение измененной базы. Недавно было показано, что модификации РНК могут быть обнаружены с помощью Oxford Nanopore direct RNA-Seq (Garalde et al., 2018).
Рисунок 2. Иллюстрация анализа эпитранскриптома с использованием антител для идентификации РНК с модификациями оснований. Поли (A) + мРНК используется для иммунопреципитации РНК с антителами, специфичными для модификации основания (например, m 6 A или m 5 C). Затем IP’ed РНК используется для создания библиотеки кДНК для высокопроизводительного секвенирования. Затем считывания сопоставляются с эталонным геномом.
Протокол конструирования библиотеки для прямой RNA-Seq был разработан для поли (A) транскриптов (Garalde et al., 2018). Шаги, участвующие в нативной РНК-Seq, проиллюстрированы на рисунке 3. Хотя одноцепочечная РНК изображена на этом рисунке, гибрид РНК-ДНК можно использовать для прямой РНК-Seq, где секвенируется только цепь РНК в гибриде. Использование гибридов РНК-ДНК может облегчить некоторые проблемы, связанные со вторичными структурами РНК, и улучшить производительность и качество последовательностей (Garalde et al., 2018). Для характеристики дрожжевого транскриптома с использованием прямой RNA-Seq использовалась одноцепочечная РНК (Garalde et al., 2018), тогда как при анализе транскриптома человека использовались гибриды РНК-ДНК (Workman et al., 2018). Транскрипты без поли (A) -хвоста также можно секвенировать путем ферментативного добавления 3′-поли (A) -хвоста. Одно из ограничений прямого RNA-Seq касается усеченных чтений. Исследования как вируса псевдобешенства (Moldován et al., 2018b), так и Saccharomyces cerevisiae (Jenjaroenpun et al., 2018) выявили усеченные чтения, особенно отсутствующие нуклеотиды на 5′-конце транскриптов. Было высказано предположение, что это могло быть связано с преждевременным высвобождением транскриптов секвенирования моторным белком (Moldován et al., 2018б). Однако более длинные транскрипты более 5 т.п.н. могут быть созданы с использованием прямой РНК-Seq (Jenjaroenpun et al., 2018). Таким образом, моторный белок не может быть основной причиной усеченных считываний. Еще одно ограничение заключается в том, что в настоящее время инструменты биоинформатики для идентификации модификации РНК встречаются редко. Tombo — единственный зарегистрированный инструмент для идентификации модифицированных нуклеотидов из ONT (Stoiber et al., 2016). Кроме того, еще предстоит разработать алгоритмы вызова оснований для большинства модификаций РНК. Недавно транскриптом семян сои ( Glycine max ) был секвенирован с использованием секвенирования MinION.Однако в этом исследовании был принят метод секвенирования кДНК, который нельзя было использовать для характеристики модификаций РНК (Fleming et al., 2018). Пока что только два исследования прямого РНК-секвенирования — одно с поли (A) + РНК дрожжей (Garalde et al., 2018) и одно с поли ( A) + РНК ( Workman et al., 2018) — были выполнены с мРНК эукариот. Интересно, что нативное секвенирование РНК поли (A) + человека выявило большое количество новых изоформ (более 65% всех обнаруженных изоформ являются новыми) (Workman et al., 2018). Авторы исследования транскриптома человека смогли оценить длину поли (A) + , аллель-специфичную экспрессию, модификации оснований (N 6 -метиладенин и инозин) в мРНК на основе прямых данных RNA-Seq (Workman et al. , 2018).
Рисунок 3. Схематическое изображение прямого секвенирования РНК с использованием Oxford Nanopore Technology. Поли (А) + мРНК из общей РНК выделяют, затем к 3′-концу поли (А) + мРНК добавляют адаптер поли (Т) и адаптер секвенирования с моторным ферментом.Затем он подвергается секвенированию на мембране с тысячами каналов нанопор, каждый из которых подключен к амперметрам, измеряющим ток, проходящий через поры. Моторный фермент взаимодействует с нанопорой на электрически устойчивой синтетической мембране, и нить РНК проходит через нанопору. Напряжение через мембрану прикладывается, и когда РНК движется через нанопоры нуклеотидных оснований, вызывает характерное изменение тока через поры, которое является уникальным для каждого нормального и модифицированного основания.Текущий выход затем используется при вызове базы. Показан пример выходного тока, когда РНК с (правое поле) или без модифицированных оснований РНК (левое окно) движется через пору.
Направления будущего
На этапе создания библиотеки Iso-Seq становится очевидно, что информация о модификации РНК будет удалена. Таким образом, обычные библиотеки Iso-Seq нельзя использовать для обнаружения модификации РНК. Помимо прямого RNA-Seq, PacBio считывает из секвенирования генома без какой-либо стадии ПЦР-амплификации, и может использоваться для обнаружения меток метилирования ДНК, таких как m 6 A, m 5 C, 5-гидроксиметилцитозин (Flusberg et al., 2010; Fang et al., 2012) и 4-метилцитозин (4mC) (Ye et al., 2016) соответственно. Бисульфитное секвенирование (BS-Seq) с использованием NGS также может обнаруживать m 5 C общегеномным способом (Krueger et al., 2012). Однако длинные считывания без ПЦР-амплификации предоставляют новые возможности для обнаружения дополнительных модификаций, которые имеют различные кинетические профили и не могут быть обнаружены с помощью технологий NGS. В A. thaliana было исследовано глобальное профилирование остатков m 6 A с использованием этого метода при разрешении одного нуклеотида (Liang et al., 2018). Секвенирование ONT может обнаруживать метилирование природного генома, о котором сообщалось у Escherichia coli (Rand et al., 2017) и людей (Simpson et al., 2017). Можно ожидать, что и PacBio, и ONT с достаточным охватом могут заменить существующие методы обнаружения метилирования, такие как обработанная бисульфитом ДНК после NGS для идентификации m 5 C (Frommer et al., 1992). При использовании обратной транскриптазы вместо ДНК-полимеразы в ZMW синтез кДНК наблюдали в режиме реального времени (Saletore et al., 2012). Кроме того, было показано, что присутствие модифицированной (например, m 6 A) в РНК изменяет кинетику включения нуклеотидов в модифицированный сайт. На основании этого было высказано предположение, что с помощью мониторинга синтеза кДНК в реальном времени в ZMWs модификации в РНК могут быть идентифицированы с использованием измененной кинетической сигнатуры (Saletore et al., 2012).
Предыдущие исследования показали, что сложно реконструировать изоформы сплайсинга и количественно оценить дифференциальную экспрессию изоформ, используя короткие считывания, полученные с помощью секвенирования второго поколения (Steijger et al., 2013; Кратц и Карнинчи, 2014). По сравнению с Illumina, длина считывания является большим преимуществом в секвенировании транскриптов кДНК Iso-Seq и прямой РНК-Seq Oxford Nanopore, которая может захватывать полные транскрипты (Wang et al., 2016). Сравнение экспрессии генов между наборами данных Illumina и MinION выявило высокий коэффициент корреляции (Seki et al., 2018), который предполагает, что MinION является полезной платформой для расчета уровня экспрессии транскриптов по количеству считываний или относительной численности РНК в виде транскриптов на млн транскриптов (TPM) (Маринов, 2017).Действительно, долгосрочное секвенирование одной молекулы кукурузы выявило тканеспецифические изоформы (Wang et al., 2016). Эти новые технологии предоставляют большие возможности и новые возможности для изучения сложных транскриптомов. Комбинация различных методов может предложить решения для преодоления недостатков NGS и PacBio / ONT (Rhoads and Au, 2015). В настоящее время IDP (Au et al., 2013) был разработан для использования длинных чтений для идентификации сложной структуры транскриптов и коротких чтений следующего поколения для количественной оценки.Этот гибридный метод может устранить ограничение для обеих технологий. Недавнее исследование показало высокую корреляцию между ONT и Illumina при количественной оценке экспрессии генов (Byrne et al., 2017). С улучшением секвенсоров (от MinION, GridION до PromethION) Oxford Nanopore direct RNA-Seq с достаточной пропускной способностью и точностью, возможно, может быть использован для выполнения количественного анализа полноразмерных изоформ на уровне всего транскриптома.
Взносы авторов
LZ, HZ, MK, KP, LG и AR написали, обсудили и отредактировали рукопись.
Финансирование
Эта работа была поддержана Национальным фондом естественных наук Китая (грант № 31570674), Национальной ключевой программой НИОКР Китая (2016YFD0600106 и 2018YFD0600101), Международным фондом научно-технического сотрудничества и обмена от Университета сельского и лесного хозяйства Фуцзянь (KXGh27016 ), Фонд естественных наук провинции Фуцзянь Китая (грант № 2018J01608) и Управление науки Министерства энергетики, Управление биологических и экологических исследований (грант №DE-SC0010733).
Заявление о конфликте интересов
Авторы заявляют, что исследование проводилось при отсутствии каких-либо коммерческих или финансовых отношений, которые могут быть истолкованы как потенциальный конфликт интересов.
Сноски
- https://github.com/PacificBiosciences/PacBioFileFormats/wiki/BAM-recipes
- https://nanoporetech.com
- https://nanoporetech.com
- https: // community.nanoporetech.com
- https://github.com/ocxtal/minialign
- https://github.com/Magdoll/cDNA_Cupcake
- https://github.com/PacificBiosciences
- https://github.com/liuxiaoxian/IsoSeq_AS_de_novo
Список литературы
Абдель-Гани, С. Э., Гамильтон, М., Якоби, Дж. Л., Нгам, П., Девитт, Н., Шилки, Ф. и др. (2016). Обзор транскриптома сорго с использованием длинных считываний одиночных молекул. Nat. Commun. 7: 11706. DOI: 10.1038 / ncomms11706
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ау, К. Ф., Себастьяно, В., Афшар, П. Т., Дуррути, Дж. Д., Ли, Л., Уильямс, Б. А. и др. (2013). Характеристика транскриптома ESC человека с помощью гибридного секвенирования. Proc. Natl. Акад. Sci. США 110, E4821 – E4830. DOI: 10.1073 / pnas.1320101110
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Бадуин, Х., Gouzy, J., Grassa, C.J., Murat, F., Staton, S.E., Cottret, L., et al. (2017). Геном подсолнечника дает представление о метаболизме масла, цветении и эволюции астерид. Природа 546, 148–152. DOI: 10.1038 / природа22380
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Байега А., Фахиминия С., Ойкономопулос С. и Рагусси Дж. (2018). «Текущие и будущие методы анализа мРНК: стремление к секвенированию одной молекулы», в Gene Expression Analysis: Methods in Molecular Biology , Vol.1783, ред. Н. Рагхавачари и Н. Гарсиа-Рейеро (Нью-Йорк, штат Нью-Йорк: Спрингер), 209–241.
Google Scholar
Beaudoing, E., Freier, S., Wyatt, J. R., Claverie, J. M., and Gautheret, D. (2000). Паттерны использования варианта сигнала полиаденилирования в генах человека. Genome Res. 10, 1001–1010. DOI: 10.1101 / gr.10.7.1001
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Блоуер М. Д., Джамбекар А., Шварц Д. С. и Тоомбс Дж. А. (2013). Комбинирование различных методов захвата мРНК для анализа транскриптома: анализ транскриптома Xenopus laevis . PLoS One 8: e77700. DOI: 10.1371 / journal.pone.0077700
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Бирн А., Боден А. Э., Олсен Х. Э., Джайн М., Коул К., Палмер Т. и др. (2017). RNAseq с длинным считыванием нанопор обнаруживает широко распространенные транскрипционные вариации среди поверхностных рецепторов индивидуальных В-клеток. Nat. Commun. 8: 16027. DOI: 10.1038 / ncomms16027
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Кэмпбелл, М.А., Хаас, Б. Дж., Гамильтон, Дж. П., Маунт, С. М., и Бьюэлл, К. Р. (2006). Комплексный анализ альтернативного сращивания риса и сравнительный анализ с Arabidopsis. BMC Genomics 7: 327. DOI: 10.1186 / 1471-2164-7-327
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Картолано М., Хюттель Б., Хартвиг Б., Рейнхардт Р. и Шнеебергер К. (2016). Обогащение библиотеки кДНК полноразмерными транскриптами для секвенирования длинного чтения smrt. PLoS One 11: e0157779.DOI: 10.1371 / journal.pone.0157779
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Чао, К., Гао, З. Ф., Чжан, Д., Чжао, Б. Г., Дун, Ф. К., Фу, К. X. и др. (2018). Динамика развития стволового транскриптома Populus. Завод Биотех. J. 17, 206–219. DOI: 10.1111 / pbi.12958
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Чао, Ю., Юань, Дж., Ли, С., Цзя, С., Хань, Л., и Сюй, Л. (2018). Анализ транскриптов и изоформ сплайсинга у клевера красного ( Trifolium pratense L.) путем секвенирования одной молекулы с длинным считыванием. bioRxiv. DOI: 10.1101 / 330977330977
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ченг Б., Фуртадо А. и Генри Р. Дж. (2017). Долговременное секвенирование транскриптома кофейных зерен показывает разнообразие полноразмерных транскриптов. Gigascience 6, 1–13. DOI: 10.1093 / gigascience / gix086
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Клавихо, Б. Дж., Вентурини, Л., Schudoma, C., Accinelli, G.G., Kaithakottil, G., Wright, J., et al. (2017). Улучшенная сборка и аннотация генома аллогексаплоидной пшеницы позволяет идентифицировать полные семейства агрономических генов и предоставляет геномные доказательства хромосомных транслокаций. Genome Res. 27, 885–896. DOI: 10.1101 / gr.217117.116
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Кук, Д. Э., Валле-Инклан, Дж. Э., Паджоро, А., Ровенич, Х., Томма, Б., и Файно, Л. (2019).Аннотации для длительного чтения: автоматическая аннотация генома эукариот на основе секвенирования кДНК методом длительного считывания. Plant Physiol. 179, 38–54. DOI: 10.1104 / стр. 18.00848
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Цуй, X., Лян, З., Шен, Л., Чжан, К., Бао, С., Гэн, Ю. и др. (2017). Метилирование 5-метилцитозиновой РНК у Arabidopsis thaliana . Мол. Завод 10, 1387–1399. DOI: 10.1016 / j.molp.2017.09.013
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Истощение, Д.П., Путханкалам, С. К., Садаока, Т., Биди, Д., Мори, Ю., Плакантонакис, Д., и др. (2018). Секвенирование нативной РНК на массивах нанопор переопределяет сложность транскрипции вирусного патогена. bioRxiv DOI: 10.1101 / 373522
CrossRef Полный текст | Google Scholar
Добин А., Дэвис К. А., Шлезингер Ф., Дренкоу Дж., Залески К., Джа С. и др. (2013). STAR: сверхбыстрый универсальный выравниватель RNA-seq. Биоинформатика 29, 15–21. DOI: 10.1093 / биоинформатика / bts635
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Доминиссини, Д., Moshitch-Moshkovitz, S., Schwartz, S., Salmon-Divon, M., Ungar, L., Osenberg, S., et al. (2012). Топология метиломов m 6 A РНК человека и мыши, выявленная с помощью m 6 A-seq. Природа 485, 201–206. DOI: 10.1038 / природа11112
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Фанг, Г., Мунера, Д., Фридман, Д. И., Мандлик, А., Чао, М. К., Банерджи, О., и др. (2012). Полногеномное картирование остатков метилированного аденина в патогенной Escherichia coli с использованием одномолекулярного секвенирования в реальном времени. Nat. Biotechnol. 30, 1232–1239. DOI: 10.1038 / NBT.2432
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Feng, J. W., Huang, S., Guo, Y. X., Liu, D., Song, J. M., Gao, J., et al. (2019). База данных по секвенированию растений ISOform (PISO): исчерпывающий репертуар полноразмерных транскриптов растений. Plant Biotechnol. J. doi: 10.1111 / pbi.13076 [Epub перед печатью].
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Филичкин, С.А., Гамильтон, М., Дхармавардхана, П. Д., Сингх, С. К., Салливан, К., Бен-Гур, А. и др. (2018). Абиотические стрессы модулируют ландшафт транскриптома тополя посредством альтернативного сплайсинга, дифференциального удержания интронов и переключения соотношения изоформ. Фронт. Plant Sci. 9: 5. DOI: 10.3389 / fpls.2018.00005
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Флеминг, М. Б., Паттерсон, Э. Л., Ривз, П. А., Ричардс, К. М., Гейнс, Т. А., и Уолтерс, К. (2018). Изучение судьбы мРНК в стареющих семенах: защита, разрушение или медленный распад? Дж.Exp. Бот. 69, 4309–4321. DOI: 10.1093 / jxb / ery215
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Флусберг, Б. А., Вебстер, Д. Р., Ли, Дж. Х., Трэверс, К. Дж., Оливарес, Э. К., Кларк, Т. А. и др. (2010). Прямое обнаружение метилирования ДНК во время секвенирования одной молекулы в реальном времени. Nat. Методы 7, 461–465. DOI: 10.1038 / nmeth.1459
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Фуассак С. и Саммет М.(2007). АСТАЛАВИСТА: динамический и гибкий анализ альтернативных событий сплайсинга в пользовательских наборах данных генов. Nucleic Acids Res. 35, W297 – W299. DOI: 10.1093 / nar / gkm311
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Фроммер М., Макдональд Л. Э., Миллар Д. С., Коллис К. М., Ватт Ф., Григг Г. В. и др. (1992). Протокол геномного секвенирования, который дает положительное отображение остатков 5-метилцитозина в отдельных цепях ДНК. Proc. Natl. Акад.Sci. США 89, 1827–1831. DOI: 10.1073 / pnas.89.5.1827
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Фу, Л., Ню, Б., Чжу, З., Ву, С., и Ли, В. (2012). CD-HIT: ускорен для кластеризации данных секвенирования следующего поколения. Биоинформатика 28, 3150–3152. DOI: 10.1093 / биоинформатика / bts565
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Гао, Ю., Ван, Х., Чжан, Х., Ван, Ю., Чен, Дж., И Гу, Л. (2017).PRAPI: конвейер анализа посттранскрипционной регуляции для Iso-Seq. Биоинформатика 34, 1580–1582. DOI: 10.1093 / биоинформатика / btx830
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Гаральде, Д. Р., Снелл, Э. А., Яхимович, Д., Сипос, Б., Ллойд, Дж. Х., Брюс, М. и др. (2018). Высоко параллельное прямое секвенирование РНК на массиве нанопор. Nat. Методы 15, 201–206. DOI: 10,1038 / Nmeth.4577
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Гонсалес-Гарай, М.Л. (2016). «Введение в секвенирование изоформ с использованием технологии тихоокеанских биологических наук (Iso-Seq)», в Transcriptomics and Gene Regulation , ed. Дж. Ву (Дордрехт: Спрингер), 141–160.
Google Scholar
Гудвин, С., Гуртовски, Дж., Эте-Сайерс, С., Дешпанде, П., Шац, М. К., и Маккомби, В. Р. (2015). Секвенирование Oxford Nanopore, исправление гибридных ошибок и сборка de novo эукариотического генома. Genome Res. 25, 1750–1756. DOI: 10.1101 / gr.1.115
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Гордон, С. П., Ценг, Э., Саламов, А., Чжан, Дж., Мэн, X., Чжао, З. и др. (2015). Распространенные полицистронные транскрипты у грибов, выявленные с помощью секвенирования одномолекулярной мРНК. PLoS One 10: e0132628. DOI: 10.1371 / journal.pone.0132628
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Haas, B.J., Papanicolaou, A., Yassour, M., Grabherr, M., Blood, P.D., Bowden, J., et al.(2013). Реконструкция последовательности транскрипта de novo из RNA-seq с использованием платформы Trinity для создания и анализа ссылок. Nat. Протоколы 8, 1494–1512. DOI: 10.1038 / nprot.2013.084
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Хаас, Б. Дж., Зальцберг, С. Л., Чжу, В., Пертеа, М., Аллен, Дж. Э., Орвис, Дж. И др. (2008). Автоматическая аннотация структуры эукариотических генов с использованием EVidenceModeler и программы для сборки сплайсированных выравниваний. Genome Biol. 9: R7. DOI: 10.1186 / GB-2008-9-1-r7
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Хихара, Ю., Камей, А., Канехиса, М., Каплан, А., Икеучи, М. (2001). ДНК-микроматричный анализ экспрессии генов цианобактерий во время акклиматизации к яркому свету. Растительная клетка 13, 793–806. DOI: 10.1105 / tpc.13.4.793
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Jenjaroenpun, P., Wongsurawat, T., Pereira, R., Patumcharoenpol, P., Ussery, D. W., Nielsen, J., et al. (2018). Полный анализ геномного и транскрипционного ландшафта с использованием секвенирования третьего поколения: тематическое исследование Saccharomyces cerevisiae CEN. ПК113-7Д. Nucleic Acids Res. 46: e38. DOI: 10.1093 / nar / gky014
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Цзи, Г., Е, В., Су, Ю., Чен, М., Хуанг, Г., и Ву, X. (2018). AStrap: идентификация альтернативного сплайсинга из последовательностей транскриптов без эталонного генома. Bioinformatics doi: 10.1093 / bioinformatics / bty1008 [Epub перед печатью].
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Джин Дж., Лю Дж., Ван Х., Вонг Л. и Чуа Н.-Х. (2013). PLncDB: база данных длинных некодирующих РНК растений. Биоинформатика 29, 1068–1071. DOI: 10.1093 / биоинформатика / btt107
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Джо, И. Х., Ли, Дж., Хонг, К. Э., Ли, Д. Дж., Бэ, В., Парк, С.G., et al. (2017). Секвенирование изоформ обеспечивает более полное представление о транскриптоме женьшеня panax. Гены 8: E228. DOI: 10.3390 / genes80
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Какрана А., Матиони С. М., Хуанг К., Хаммонд Р., Вандивье Л., Патель П. и др. (2018). Выращивайте 24-нуклеотидные репродуктивные фазиРНК из внутримолекулярных дуплексных мРНК в различных однодольных. Geome Res. 28, 1333–1344. DOI: 10.1101 / gr.228163.117
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ким, Н.Х., Джаякоди, М., Ли, С.С., Чой, Б.С., Янг, В., Ли, Дж. И др. (2018). Геном и эволюция теневыносливой лекарственной травы Panax ginseng. Plant Biotechnol. J. 16, 1904–1917. DOI: 10.1111 / pbi.12926
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Корен, С., Шац, М. К., Валенц, Б. П., Мартин, Дж., Ховард, Дж. Т., Ганапати, Г. и др. (2012). Гибридная коррекция ошибок и сборка de novo считываний секвенирования одной молекулы. Nat. Biotechnol. 30, 693–700. DOI: 10.1038 / NBT.2280
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Левен, М. Дж., Корлах, Дж., Тернер, С. У., Фоке, М., Крейгхед, Х. Г. и Уэбб, У. У. (2003). Волноводы с нулевой модой для анализа одиночных молекул при высоких концентрациях. Наука 299, 682–686. DOI: 10.1126 / science.1079700
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ли, Х., Ван, Дж., Мор, Г., и Склар, Дж. (2008). Слияние неопластических генов имитирует транс-сплайсинг РНК в нормальных клетках человека. Science 321, 1357–1361. DOI: 10.1126 / science.1156725
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ли, Дж., Харата-Ли, Ю., Дентон, М. Д., Фенг, К., Ратиен, Дж. Р., Ку, З., и др. (2017). Реконструкция полноразмерного транскриптома Astragalusmbranaceus без использования длинного эталонного генома позволяет выявить варианты транскриптов, участвующие в биосинтезе биологически активных соединений. Cell Discov. 3: 17031. DOI: 10.1038 / celldisc.2017.31
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ли, С., Ямада, М., Хан, X., Олер, У., и Бенфей, П. Н. (2016). Карта экспрессии корня Arabidopsis с высоким разрешением показывает альтернативный сплайсинг и регуляцию lincRNA. Dev. Ячейка 39, 508–522. DOI: 10.1016 / j.devcel.2016.10.012
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ли Ю., Дай К., Ху К., Лю З. и Канг К. (2017). Глобальная идентификация альтернативного сплайсинга посредством сравнительного анализа последовательности РНК на основе SMRT и Illumina в клубнике. Плант Дж. 90, 164–176. DOI: 10.1111 / tpj.13462
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Liang, Z., Shen, L., Cui, X., Bao, S., Geng, Y., Yu, G., et al. (2018). Метилирование N6-аденина ДНК у Arabidopsis thaliana . Dev. Ячейка 45, 406–416.e3. DOI: 10.1016 / j.devcel.2018.03.012
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Лю С., Цай В.-Х., Дин Ю., Чен Р., Фанг З., Хо З. и др. (2015). Всесторонняя оценка алгоритмов обнаружения слитных транскриптов и мета-вызывающей стороны для объединения наиболее эффективных методов в данных парных последовательностей РНК. Nucleic Acids Res. 44: e47. DOI: 10.1093 / nar / gkv1234
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Лю X., Мэй В., Солтис П. С., Солтис Д. Э. и Барбазук В. Б. (2017). Обнаружение альтернативно сплайсированных изоформ транскриптов из одномолекулярных последовательностей с длинным считыванием без эталонного генома. Мол. Ecol. Ресурс. 17, 1243–1256. DOI: 10.1111 / 1755-0998.12670
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Луо, Г.-З., Маккуин, А., Чжэн, Г., Дуан, Х., Доре, Л.С., Лу, З. и др. (2014). Уникальные особенности метилома m 6 A Arabidopsis thaliana . Nat. Commun. 5: 5630. DOI: 10.1038 / ncomms6630
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Махер, К. А., Паланисами, Н., Бреннер, Дж. К., Цао, X., Кальяна-Сундарам, С., Луо, С. и др. (2009). Обнаружение химерного транскрипта путем секвенирования транскриптома с парным концом. Proc. Natl. Акад. Sci. НАС.А. 106, 12353–12358. DOI: 10.1073 / pnas.00106
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Марше, К., Лекомпт, Л., Да Силва, К., Круод, К., Аури, Дж. М., Николас, Дж. И др. (2018). Кластеризация de novo по гену длинных чтений из данных транскриптомики. Nucleic Acids Res. 42: e2. DOI: 10.1093 / nar / gky1834
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Мацумура, Х., Нирасава, С., и Тераучи, Р. (1999).Профилирование транскриптов в проростках риса ( Oryza sativa L.) с использованием серийного анализа экспрессии генов (SAGE). Plant J. 20, 719–726. DOI: 10.1046 / j.1365-313X.1999.00640.x
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Мейер, К. Д., Салеторе, Ю., Зумбо, П., Элементо, О., Мейсон, К. Э., и Джеффри, С. Р. (2012). Всесторонний анализ метилирования мРНК выявляет обогащение 3′-НТО и почти стоп-кодонами. Cell 149, 1635–1646.DOI: 10.1016 / j.cell.2012.05.003
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Минио А., Массоннет М., Вондрас А., Фигероа-Бальдерас Р., Бланко-Улате Б. и Канту Д. (2018). Аннотации в масштабе изоформ и профили экспрессии транскриптома Каберне Совиньон с использованием одномолекулярного секвенирования полноразмерной кДНК. bioRxiv doi: 10.1534 / g3.118.201008
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Миноче, А.E., Dohm, J.C., Schneider, J., Holtgrawe, D., Viehover, P., Montfort, M., et al. (2015). Использование секвенирования одномолекулярных транскриптов для предсказания эукариотических генов. Genome Biol. 16: 184. DOI: 10.1186 / s13059-015-0729-7
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Молдован, Н., Томбац, Д., Сеч, А., Чабай, З., Балаж, З., Кис, Э., и др. (2018a). Секвенирование третьего поколения выявляет обширный полицистронизм и перекрытие транскрипции в бакуловирусе. Sci. Отчет 8: 8604. DOI: 10.1038 / s41598-018-26955-8
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Молдован, Н., Томбац, Д., Сеч, А., Чабай, З., Снайдер, М., и Болдогкки, З. (2018b). Мультиплатформенный подход к секвенированию позволяет выявить новый профиль транскриптома у вируса псевдобешенства. Фронт. Microbiol. 8: 2708. DOI: 10.3389 / fmicb.2017.02708
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Мортазави, А., Уильямс, Б.А., Макку, К., Шеффер, Л., и Уолд, Б. (2008). Картирование и количественная оценка транскриптомов млекопитающих с помощью RNA-Seq. Nat. Методы 5: 621. DOI: 10.1038 / nmeth.1226
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Мортон, Т., Петрика, Дж., Коркоран, Д. Л., Ли, С., Винтер, К. М., Карда, А. и др. (2014). Анализ парных концов сайтов начала транскрипции у Arabidopsis выявляет специфичные для растений сигнатуры промоторов. Растительная клетка 26, 2746–2760.DOI: 10.1105 / tpc.114.125617
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Нагасаки, Х., Арита, М., Нисидзава, Т., Сува, М., и Гото, О. (2006). Автоматическая классификация альтернативного сплайсинга и инициации транскрипции, а также построение визуальной базы данных классифицированных паттернов. Биоинформатика 22, 1211–1216. DOI: 10.1093 / биоинформатика / btl067
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Нам, Д. К., Ли, С., Zhou, G., Cao, X., Wang, C., Clark, T., et al. (2002). Праймер олиго (dT) с высокой частотой генерирует усеченные кДНК за счет внутреннего прайминга поли (A) во время обратной транскрипции. Proc. Natl. Акад. Sci. США 99, 6152–6156. DOI: 10.1073 / pnas.092140899
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ni, T., Corcoran, D. L., Rach, E. A., Song, S., Spana, E. P., Gao, Y., et al. (2010). Стратегия парного секвенирования для картирования сложного ландшафта инициации транскрипции. Nat. Методы 7: 521. DOI: 10.1038 / Nmeth.1464
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Нин, Г., Ченг, X., Луо, П., Лян, Ф., Ван, З., Ю, Г. и др. (2017). Гибридное секвенирование и поиск карт (HySeMaFi): дополнительные стратегии для всестороннего расшифровки сплайсинга и экспрессии генов в организмах без эталонного генома. Sci. Отчет 7: 43793. DOI: 10.1038 / srep43793
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Pootakham, W., Sonthirod, C., Naktang, C., Ruang-Areerate, P., Yoocha, T., Sangsrakru, D., et al. (2017). Гибридная сборка de novo генома каучукового дерева выявляет доказательства палеотетраплоидии у видов гевеи. Sci. Отчет 7: 41457. DOI: 10.1038 / srep41457
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Рамскельд, Д., Луо, С., Ван, Й.-К., Ли, Р., Дэн, К., Фаридани, О. Р. и др. (2012). Полноразмерная мРНК-Seq из одноклеточных уровней РНК и отдельных циркулирующих опухолевых клеток. Nat. Biotechnol. 30, 777–782. DOI: 10.1038 / NBT.2282
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Рэнд, А. К., Джайн, М., Эйзенга, Дж. М., Массельман-Браун, А., Олсен, Х. Э., Акесон, М., и др. (2017). Картирование метилирования ДНК с помощью высокопроизводительного секвенирования нанопор. Nat. Методы 14, 411–413. DOI: 10.1038 / nmeth.4189
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Салюторе, Ю., Мейер, К., Корлах, Дж., Вильфан, И. Д., Джеффри, С., Мейсон, К. Э. (2012). Рождение эпитранскриптома: расшифровка функции модификаций РНК. Genome Biol. 13: 175. DOI: 10.1186 / GB-2012-13-10-175
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Салмела, Л., Вальве, Р., Соперники, Э., и Укконен, Э. (2016). Точная самокоррекция ошибок при длинных чтениях с использованием графиков де Брейна. Биоинформатика 33, 799–806. DOI: 10.1093 / биоинформатика / btw321
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Седлажецк, Ф.Дж., Ли, Х., Дарби, К. А. и Шац, М. К. (2018). Проникновение в темную материю: биоинформатика дальнего секвенирования и картирования. Nat. Преподобный Жене. 19, 329–346. DOI: 10.1038 / s41576-018-0003-4
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Секи, М., Кацумата, Э., Судзуки, А., Сереваттанавут, С., Сакамото, Ю., Мидзусима-Сугано, Дж. И др. (2018). Оценка и применение RNA-Seq с помощью MinION. DNA Res. 26, 55–65. DOI: 10.1093 / dnares / dsy038
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Шен, С., Park, J. W., Lu, Z.-X., Lin, L., Henry, M. D., Wu, Y. N., et al. (2014). rMATS: надежное и гибкое обнаружение дифференциального альтернативного сплайсинга на основе данных репликации RNA-Seq. Proc. Natl. Акад. Sci. США 111, E5593 – E5601. DOI: 10.1073 / pnas.14111
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Шепард, П. Дж., Чой, Э.-А., Лу, Дж., Фланаган, Л. А., Хертель, К. Дж., И Ши, Ю. (2011). Сложный и динамичный ландшафт полиаденилирования РНК, выявленный с помощью PAS-Seq. РНК 17, 761–772. DOI: 10.1261 / rna.2581711
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Шерстнев А., Дык, К. Л., Коул, К., Захараки, В., Хорниик, К., Озсолак, Ф., и др. (2012). Прямое секвенирование РНК Arabidopsis thaliana выявляет закономерности расщепления и полиаденилирования. Nat. Struct. Мол. Биол. 19, 845–852. DOI: 10.1038 / nsmb.2345
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Симпсон, Дж. Т., Уоркман, Р. Э., Зузарте, П., Дэвид, М., Дурси, Л., и Тимп, В. (2017). Обнаружение метилирования цитозина ДНК с помощью секвенирования нанопор. Nat. Методы 14, 407–410. DOI: 10.1038 / nmeth.4184
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Слободин Б., Хан Р., Кальдероне В., Вриелинк Дж., Лоайза-Пуч Ф., Элкон Р. и др. (2017). Транскрипция влияет на эффективность трансляции мРНК посредством котранскрипционного метилирования N6-аденозина. Ячейка 169: e312.DOI: 10.1016 / j.cell.2017.03.031
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Смит, А. М., Джайн, М., Малруни, Л., Гаральд, Д. Р., и Акесон, М. (2017). Чтение канонических и модифицированных нуклеотидов в рибосомной РНК 16S с использованием прямого секвенирования РНК нанопор. bioRxiv. DOI: 10.1101 / 132274
CrossRef Полный текст | Google Scholar
Steijger, T., Abril, J. F., Engstrom, P. G., Kokocinski, F., Hubbard, T. J., Guigo, R., et al. (2013).Оценка методов реконструкции транскриптов для RNA-seq. Nat. Методы 10, 1177–1184. DOI: 10.1038 / nmeth.2714
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Стойбер, М. Х., Квик, Дж., Иган, Р., Ли, Дж. Э., Сельникер, С. Е., Нили, Р. и др. (2016). Идентификация de novo модификаций ДНК благодаря обработке сигналов нанопор, управляемой геномом. bioRxiv. DOI: 10.1101 / 094672
CrossRef Полный текст | Google Scholar
Вс, м.-Y., Li, J.-Y., Li, D., Huang, F.-J., Wang, D., Li, H., et al. (2018). Полноразмерное секвенирование транскриптома и анализ модульной организации паттерна экспрессии генов, связанных с нарингином / неоэриоцитрином, в Drynaria roosii . Physiol растительных клеток. 59, 1398–1414. DOI: 10.1093 / pcp / pcy072
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Танака Т., Коянаги К. О. и Ито Т. (2009). Высоко диверсифицированная молекулярная эволюция нисходящих стартовых сайтов транскрипции у риса и Arabidopsis. Plant Physiol. 149, 1316–1324. DOI: 10.1104 / стр.108.131656
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Тиан Б., Ху Дж., Чжан Х. и Лутц К. С. (2005). Масштабный анализ полиаденилирования мРНК генов человека и мыши. Nucleic Acids Res 33, 201–212. DOI: 10.1093 / nar / gki158
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Тиан, Дж., Фэн, С., Лю, Ю., Чжао, Л., Тиан, Л., Ху, Ю. и др. (2018).Одномолекулярное долгосрочное секвенирование Zanthoxylum bungeanum Maxim. Транскриптом: идентификация генов, связанных с ароматом. Лес 9: 765. DOI: 10.3390 / f
65
CrossRef Полный текст | Google Scholar
Tilgner, H., Jahanbani, F., Blauwkamp, T., Moshrefi, A., Jaeger, E., Chen, F., et al. (2015). Всесторонний анализ транскриптома с использованием синтетического секвенирования с длительным считыванием показывает молекулярную ассоциацию отдаленных событий сплайсинга. Nat. Biotechnol. 33, 736–742. DOI: 10.1038 / NBT.3242
PubMed Аннотация | CrossRef Полный текст | Google Scholar
ВанБурен, Р., Брайант, Д., Эджер, П. П., Тан, Х., Берджесс, Д., Чаллабатула, Д. и др. (2015). Одномолекулярное секвенирование устойчивой к высыханию травы Oropetium thomaeum . Природа 527, 508–511. DOI: 10.1038 / nature15714
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ван, Б., Ценг, Э., Регульски, М., Кларк, Т.A., Hon, T., Jiao, Y., et al. (2016). Раскрытие сложности транскриптома кукурузы путем долгого чтения одной молекулы. Nat. Commun. 7: 11708. DOI: 10.1038 / ncomms11708
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ван, Х.-Л. В., Чеканова Дж. А. (2017). «Длинные некодирующие РНК в растениях», в Long Noncode RNA Biology: Advances in Experimental Medicine and Biolog , ed. М. Рао (Сингапур: Springer), 133–154. DOI: 10.1007 / 978-981-10-5203-3_5
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ван, М., Wang, P., Liang, F., Ye, Z., Li, J., Shen, C., et al. (2018). Глобальный обзор альтернативного сращивания аллополиплоидного хлопка: ландшафт, сложность и регулирование. New Phytol. 217, 163–178. DOI: 10.1111 / nph.14762
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ван, К., и Рио, Д. К. (2018). JUM — это вычислительный метод для всестороннего анализа без аннотаций альтернативных паттернов сплайсинга пре-мРНК. Proc. Natl. Акад. Sci. США 115, E8181 – E8190.DOI: 10.1073 / pnas.1806018115
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Wang, T., Wang, H., Cai, D., Gao, Y., Zhang, H., Wang, Y., et al. (2017). Всестороннее профилирование ассоциированного с корневищем альтернативного сплайсинга и альтернативного полиаденилирования у мозобамбука ( Phyllostachys edulis ). Plant J. 91, 684–699. DOI: 10.1111 / tpj.13597
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ван, X., Сюй, Y., Zhang, S., Cao, L., Huang, Y., Cheng, J., et al. (2017). Геномный анализ примитивных, диких и культурных цитрусовых дает представление о бесполом размножении. Nat. Genet. 49, 765–772. DOI: 10,1038 / нг.3839
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Weirather, J. L., Afshar, P. T., Clark, T. A., Tseng, E., Powers, L. S., Underwood, J. G., et al. (2015). Характеристика генов слияния и значительно экспрессируемых изоформ слияния при раке молочной железы путем гибридного секвенирования. Nucleic Acids Res. 43: e116. DOI: 10.1093 / nar / gkv562
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Weirather, J. L., De Cesare, M., Wang, Y., Piazza, P., Sebastiano, V., Wang, X. J., et al. (2017). Всестороннее сравнение Pacific Biosciences и Oxford Nanopore Technologies и их приложений для анализа транскриптомов. F1000Res 6: 100. DOI: 10.12688 / f1000research.10571.2
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Уоркман, Р.E., Tang, A.D., Tang, P. S., Jain, M., Tyson, J. R., Zuzurate, P. C., et al. (2018). Секвенирование нативной РНК нанопорами поли (А) транскриптома человека. bioRxiv. DOI: 10.1101 / 459529
CrossRef Полный текст | Google Scholar
Ву Б., Суо Ф., Лей В. и Гу Л. (2014). Комплексный анализ альтернативного сплайсинга Digitalis purpurea с помощью специфической для цепи RNA-Seq. PLoS One 9: e106001. DOI: 10.1371 / journal.pone.0106001
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ву, Дж., Maehara, T., Shimokawa, T., Yamamoto, S., Harada, C., Takazaki, Y., et al. (2002). Полная карта транскриптов риса, содержащая 6591 сайт меток экспрессированной последовательности. Растительная клетка 14, 525–535. DOI: 10.1105 / tpc.010274
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Сяо, В., Адхикари, С., Дахал, Ю., Чен, Ю.С., Хао, Ю.Дж., Сан, Б.Ф. и др. (2016). Ядерный m (6) A-ридер YTHDC1 регулирует сплайсинг мРНК. Мол. Ячейка 61, 507–519. DOI: 10.1016 / j.molcel.2016.01.012
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Xie, S. Q., Han, Y., Chen, X. Z., Cao, T. Y., Ji, K. K., Zhu, J., et al. (2018). ISOdb: обширная база данных полноразмерных изоформ, созданная Iso-Seq. Внутр. Дж. Геномикс 2018: 9207637. DOI: 10.1155 / 2018/9207637
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Xu, Q., Zhu, J., Zhao, S., Hou, Y., Li, F., Tai, Y., et al. (2017). Профилирование транскриптома с использованием метода прямого секвенирования одной молекулы РНК для глубокого понимания генов вторичных путей метаболизма Camellia sinensis . Фронт. Plant Sci. 8: 1205. DOI: 10.3389 / fpls.2017.01205
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Xu, Z., Peters, R.J., Weirather, J., Luo, H., Liao, B., Zhang, X., et al. (2015). Полноразмерные последовательности транскриптомов и варианты сплайсинга, полученные с помощью комбинации платформ для секвенирования, примененных к различным тканям корня шалфея Salvia miltiorrhiza и биосинтеза таншинона. Plant J. 82, 951–961. DOI: 10.1111 / tpj.12865
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Ян, Л., Цзинь, Ю., Хуанг, В., Сан, К., Лю, Ф., и Хуанг, X. (2018). Полноразмерные последовательности транскриптома эфемерного растения Arabidopsis pumila позволяют понять динамику экспрессии генов во время непрерывного солевого стресса. BMC Genomics 19: 717. DOI: 10.1186 / s12864-018-5106-y
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Е, П., Луан, Ю., Чен, К., Лю, Ю., Сяо, К., и Се, З. (2016). MethSMRT: комплексная база данных ДНК N6-метиладенина и N4-метилцитозина, созданная путем одномолекулярного секвенирования в реальном времени. Nucleic Acids Res. 45, D85 – D89. DOI: 10.1093 / nar / gkw950
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Чжан Б., Лю Дж., Ван Х. и Вэй З. (2018). Полноразмерное секвенирование РНК выявило уникальный состав транскриптомов бермудских трав. Plant Physiol. Biochem. 132, 95–103. DOI: 10.1016 / j.plaphy.2018.08.039
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Чжан, Х., Ван, Х., Чжу, К., Гао, Ю., Ван, Х., Zhao, L., et al. (2018). Транскриптомная характеристика проростков мозо-бамбука ( Phyllostachys edulis ) в ответ на внесение экзогенного гиббереллина. BMC Plant Biol. 18: 125. DOI: 10.1186 / s12870-018-1336-z
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Zhang, G., Guo, G., Hu, X., Zhang, Y., Li, Q., Li, R., et al. (2010). Глубокое секвенирование РНК при разрешении одной пары оснований показывает высокую сложность транскриптома риса. Genome Res. 20, 646–654. DOI: 10.1101 / gr.100677.109
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Zhang, Y., Gu, L., Hou, Y., Wang, L., Deng, X., Hang, R., et al. (2015). Интегративный полногеномный анализ показывает, что HLP1, новый связывающий РНК белок, регулирует цветение растений, воздействуя на альтернативное полиаденилирование. Cell Res. 25, 864–876. DOI: 10.1038 / cr.2015.77
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Чжао, Х., Гао, З., Ван, Л., Ван, Дж., Ван, С., Фей, Б. и др. (2018). Эталонный геном на уровне хромосом и атлас альтернативного сплайсинга мозобамбука (Phyllostachys edulis). Gigascience 7: giy115. DOI: 10.1093 / gigascience / giy1115
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Чжоу Р., Мошгабади Н. и Адамс К. Л. (2011). Обширные изменения в альтернативных схемах сплайсинга после аллополиплоидии в природных и повторно синтезированных полиплоидах. Proc.Natl. Акад. Sci, США, 108, 16122–16127. DOI: 10.1073 / pnas.1109551108
PubMed Аннотация | CrossRef Полный текст | Google Scholar
GitHub — PacificBiosciences / IsoSeq: IsoSeq3 — Масштабируемое обнаружение изоформ De Novo из одномолекулярного пакета PacBio читает
Масштабируемое открытие изоформ De Novo
IsoSeq v3 содержит новейшие инструменты для идентификации транскриптов в
Данные секвенирования одной молекулы PacBio.
Начиная с SMRT Link v6.0.0, эти инструменты обеспечивают работу
Приложение анализа на основе графического интерфейса пользователя IsoSeq .Составной рабочий процесс из существующих инструментов и алгоритмов в сочетании с
новый метод кластеризации, позволяющий обрабатывать постоянно растущий урожай PacBio
машины с производительностью, аналогичной IsoSeq версий 1 и 2.
Начиная с версии 3.4, поддержка UMI и дедупликации на основе штрих-кода ячейки.
был добавлен.
Наличие
Последнюю версию можно установить с помощью пакета bioconda isoseq3 .
Пожалуйста, посетите нашу официальную страницу pbbioconda
для получения информации об установке, поддержке, лицензии, авторских правах и отказе от ответственности.
Документация рабочего процесса
История изменений
- 3.4.0
- Версия SMRT Link 10.0.0
- Добавьте поддержку UMI и обработки штрих-кода ячеек, добавив тег
dedup - Добавьте
Уточнить --min-rqдля поддержки фильтрации RQ для нефильтрованных.reads.bam
- 3.3.0
- 3.2.2
- Fix
polishне генерирует вывод fasta / q.Эта ошибка появилась в версии 3.2.0
.
- Fix
- 3.2.1
- Исправить одноразовую ошибку индекса gff в обрушении
- Мы удалили неявные зависимости из рецепта биоконды. При необходимости установите
pbccs,limaиpbcoretools.
- Исправить одноразовую ошибку индекса gff в обрушении
- 3.2.0
-
polishбольше не поддерживает наборы данных RS II! - Добавить
свернутьшаг для выровненной расшифровки ввода BAM - Включить рабочий процесс только для CCS
кластер --use-qvs - Добавить
Уточнить --min-polya-length - Добавить
cluster --singletonsдля вывода некластеризованных FLNC; возможные артефакты при подготовке образцов! - Исправить ошибки Minimap2.Выходы могут немного измениться.
-
- 3.1.2
- Уменьшить
полировкаОбъем памяти
- Уменьшить
- 3.1.1
- Фиксация края корпуса, где полироль
- Улучшить
отполировать время выполнениядля крупномасштабных наборов данных (> 1 млн CCS) - Улучшить
полирольКачество результата
- Фиксация края корпуса, где полироль
- 3.1.0
- Мы передали на аутсорсинг удаление хвостов поли (A) и обнаружение конкатемеров в новый инструмент.
позвонил, доработать.Ваши индивидуальные праймеры.fastaиспользуется на этом этапе для обнаружения
конкатемеры.
- Мы передали на аутсорсинг удаление хвостов поли (A) и обнаружение конкатемеров в новый инструмент.
FAQ
Где рабочий процесс начинается с неполированных чтений CCS?
Чтобы упростить, унифицировать и обеспечить соответствие требованиям будущего Iso-Seq, мы решили удалить документацию.
начиная с неотшлифованных прочтений CCS. С постоянно увеличивающимся количеством полимераз читать
продолжительности и усовершенствованиях CCS, в будущем рекомендуется создать
полировка CCS считывается первой, поэтому окончательная полировка стенограммы становится необязательной.
Почему IsoSeq v3, а не установленная версия 1 или 2?
Постоянно увеличивающаяся пропускная способность системы Sequel привела к необходимости
масштабируемое программное решение, способное обрабатывать миллионы операций чтения CCS, в то время как
сохранение чувствительности и точности. Внутренние тесты показали, что
IsoSeq v3 на порядки быстрее, чем используемые в настоящее время решения и
Атрибуты SQANTI IsoSeq v3 и выше
количество идеально аннотированных изоформ:
Дополнительное преимущество, один двоичный файл Linux, не требующий зависимостей.
Почему количество транскриптов намного меньше с IsoSeq v3?
Несмотря на то, что мы также наблюдаем меньше полированных транскриптов с IsoSeq v3 ,
общее качество намного выше. Большинство стенограмм низкого качества теряются в
шаг демультиплексирования. Isoseq v1 / 2 classify слишком расслаблен и не фильтрует
нежелательные молекулы до удовлетворительного уровня. Фактически, лима звонков на место и
эффективно удаляет большинство молекул, которые ошибочно помечены, как в двух 5 ‘или двух
3 ‘праймеры.Только правильная пара праймеров 5 ‘и 3’ позволяет идентифицировать полноразмерный
стенограмма и ее ориентация.
Я не могу найти
классифицируйте шаг
Начиная с версии 3.1, функция classify разделена на два инструмента.
Удаление (штрих-кодов) праймеров выполняется с помощью стандартного демультиплексирования PacBio.
инструмент лима . Lima не удаляет поли (A) хвосты и не обнаруживает конкатемеры.
Для этого isoseq Уточнить генерирует чтения FLNC.
Для версии 3.0 удаление хвоста поли (A) и обнаружение конкатемеров выполняется в
isoseq кластер
У моего образца поли (A) хвосты, как их удалить?
Используйте --require-polya для isoseq Уточнить .
Фильтр для чтения FL с поли (A) хвостом.
минимум с 20 парами оснований и удаляет идентифицированный хвост.
Сколько времени займет обработка моих данных?
Нет функции ETA. В зависимости от типа образца полный транскриптом
или целевой амплификации, время выполнения варьируется.Такое же количество чтений из
весь образец транскриптома может завершить кластеризацию за считанные минуты, тогда как один
Амплификация гена транскриптов размером 10kb может занять пару часов.
Какой алгоритм кластеризации используется?
В отличие от своих предшественников, IsoSeq v3 не использует NP-hard clique
поиск, но использует стратегию иерархического выравнивания с O (N * log (N)) .
Недавние достижения в быстром согласовании длинных чтений делают этот подход
достижимый.
Сколько операций чтения CCS используется для представления неотшлифованной последовательности кластера?
Кластер использует до 10 операций чтения CCS для генерации необработанного консенсуса кластера.
Сколько субпотоков используется для полировки?
Polish использует до 60 субпотоков для полировки кластерного консенсуса.
Когда два чтения сгруппированы?
IsoSeq v3 считает, что два чтения происходят из одного и того же транскрипта, если они встречаются
следующие критерии:
Нет верхнего предела количества пропусков.
Объяснение тегов BAM
Используются следующие теги BAM:
-
ibСводка штрих-кода: тройки, разделенные точкой с запятой, каждая тройка содержит два индекса штрих-кода и счетчики ZMW, разделенные запятой.Пример:0,1,20; 0,3,5 -
icСумма количества проходов от всех ZMW, использованных для достижения консенсуса -
imZMW-имена, связанные с этой изоформой -
-Число ZMW, связанных с этой изоформой -
itСписок штрих-кодов / UMI, вырезанных во время тега -
изМаксимальное количество подпиток, используемых для полировки -
rqПрогнозируемая точность для полированной изоформы -
XAПорядоктегнаименования -
XCпоследовательность штрих-кодатег -
XGPacBio’sGGGСуффикс UMIтег -
XMПоследовательность UMIтег -
XOПоследовательность выступовметка
Значения качества ограничены 93 .
Какие конструкции SMRTbell возможны?
PacBio поддерживает три различных дизайна SMRTbell для библиотек IsoSeq.
Во всех конструкциях транскрипты помечены асимметричными праймерами,
в то время как поли (A) хвост не является обязательным. По желанию могут быть добавлены штрих-коды.
Бинарный файл не работает в моей системе Linux!
Двоичные файлы требуют поддержки ЦП SSE4.1 ; Процессоры после 2008 года (Penryn) включают его.
ОТКАЗ ОТ ОТВЕТСТВЕННОСТИ
НАСТОЯЩИЙ ВЕБ-САЙТ, СОДЕРЖАНИЕ И ВСЕ УСЛУГИ, СВЯЗАННЫЕ С САЙТОМ, ВКЛЮЧАЯ ЛЮБЫЕ ДАННЫЕ, ПРЕДОСТАВЛЯЮТСЯ «КАК ЕСТЬ», СО ВСЕМИ ОШИБКАМИ, БЕЗ ЗАЯВЛЕНИЙ ИЛИ ПОДРАЗУМЕВАЕМЫХ ГАРАНТИЙ, ВКЛЮЧАЯ, НО НЕ ОГРАНИЧИВАЯСЬ НИКАКИМИ ГАРАНТИЯМИ ТОВАРНОЙ ПРИГОДНОСТИ, УДОВЛЕТВОРИТЕЛЬНОГО КАЧЕСТВА, НЕДОСТАТОЧНОСТИ НАРУШЕНИЯ ИЛИ ПРИГОДНОСТИ ДЛЯ КОНКРЕТНОЙ ЦЕЛИ.ВЫ НЕСЕТЕ ПОЛНУЮ ОТВЕТСТВЕННОСТЬ И РИСКИ ЗА ИСПОЛЬЗОВАНИЕ ДАННОГО САЙТА, ВСЕХ УСЛУГ, СВЯЗАННЫХ С САЙТОМ, И ЛЮБЫХ САЙТОВ ИЛИ ПРИЛОЖЕНИЙ ТРЕТЬИХ ЛИЦ.

 фунтов стерлингов — годовой вклад стандартов в экономику Великобритании
фунтов стерлингов — годовой вклад стандартов в экономику Великобритании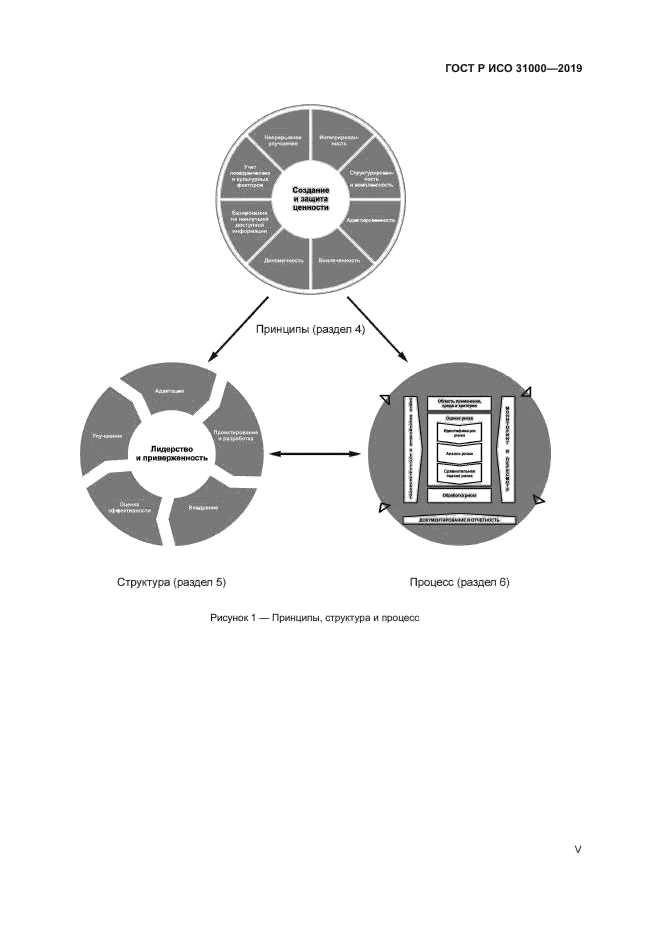
 16.1.5)
16.1.5)